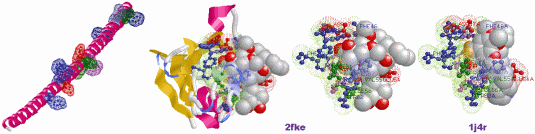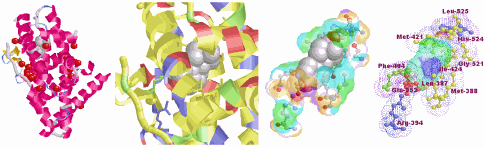
作成図例1 1ere-Chain AのSITE表示:拡大(左2つは全体表示,右2つはLigand結合部位;SITE部位は1qkuのSITE情報と同配列を抽出)
※Ligandは女性ホルモン17β-estradiol → 環境ホルモンの3D-QSAR紹介,分子と分子の相互作用,ステロイドホルモンの生合成と代謝
※本コンテンツはデータが増えて使いにくくなったため,フレーム版に更新し,本旧版でのデータ追加は停止していますので,今後はフレーム版をご利用ください。以下の右欄のLigand+SITE表示で,PDBデータ内の“SITE”行に記載されているLigand結合部位(活性部位)だけを拡大して参照できます。全体表示(ただしPDB部分データリストに掲載しているもの;オリジナルデータへは同リストからリンク)ボタンと交互に押せばタンパク質中の位置などを確認可能です。全体表示後に,SITE選択ボタンを押せば,左欄のProtein選択・DNA/RNA選択・Ligand選択・全選択と同様に選択部分だけ表示変更などができます。
なお,#0を付したデータは本ページ独自の表示を可能にするために,原子団No.を一部変更しています。
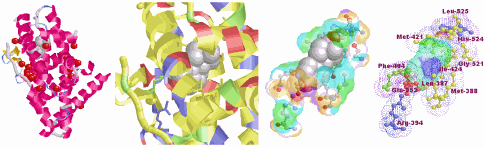
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||