| Ligand+SITE表示 | 全体表示 | SITE選択 |
| | |
| | |
| | |
| (略 → オリジナルPDB) | |
| | |
| | |
| | |
| | |
| | |
| (略 → オリジナルPDB) | |
| | |
| |
| |
| 1aay *$ | A | B | C |
| 1acm/L+S #1 | (略 → オリジナルPDB) | |
| 1aco/L+S (同配列=1ami・1amj・7acn・8acnなど;何れもHOH除く) | (略 → オリジナルPDB) | SITE全体
FS4 | HOH | TRA(L) |
| 1adb/L(DMA)+S #0,1,## | 1adbのChain A #0 | |
| 1adb/L(NAA)+S #0,## | |
| 1akb/L+S (1akcと同配列)#1 | (略 → オリジナルPDB) | |
| 1akc/L+S (1akb,1arhと同配列;類似配列=1ahy)#1 | (略 → オリジナルPDB) | |
◎参考教材1 1akd,1nooの各HEM+CAM重ね合わせデータ [P450]
…1akdは1S-camphor(ACAM),1nooは5-exo-hydroxycamphor
…Protein選択による表示変更無効 | 1akd
1noo #0 | 1akd: HEM | CAM
1noo: HEM | CAM |
| 6ald/L(CAT)+S * | (略 → オリジナルPDB) | |
| 1aq1/L+S (1ckpと類似配列) | 1aq1 | |
| 1arg/L+S (A鎖:19残基・H鎖:4残基;類似L&S=1ahe,1ahx,1akc,1akb,1arh,1ari)#0 | 1argのChain A*・B(site) #0 | |
| 4at1/L+S [ATP] | 4at1のChain B | |
| 1avd/L+S (1ave〔Ligandなし〕と同配列,1bibと同Ligand) | 1avdのChain A | |
| 1ax4/L(LPA)+S #0,1 | (略 → オリジナルPDB) | |
| 1axs/L+S #0 | 1axsのChain L・H #0 | |
| 1az1/L(ALR×2)+S (SITE記載なし;1us0と同配列選択) | 1az1 | |
| 1ben/L+S #0,1 | 1benのChain B・C・D * #0 | |
| 1bkf/(L+S) [FK506,タクロリムス] (2fkeと同配列抽出;ただし2fkeのHis87がVal87) | 1bkf | |
| 1bib/L(CAT)+S (1avdと同Ligand) | (略 → オリジナルPDB) | |
| 1bo4/L+S | 1bo4のChain A | |
| 1brp/L+S (1hbpと同配列) | (略 → オリジナルPDB) | |
| 1bsx/L+S ## | 1bsxのChain A・X | |
| 1btc/L+S * [α-シクロデキストリン] | 1btc | |
| 2btf/L(NUC)+S [ATP] | (略 → オリジナルPDB) | |
| 1btw/L(BIN)+S (1btx,1btyと同配列;他に1btz) | (略 → オリジナルPDB) | |
| 1btx/L(BLY)+S (1btw,1btyと同配列) | (略 → オリジナルPDB) | |
| 1bty/L(BIN)+S (1btw,1btxと同配列) | (略 → オリジナルPDB) | |
| 1bzf/L+S | 1bzfのModel 1 | |
| 1c8m/L+S | 1c8mのModel 1 | |
| 1ca2/L+S+近傍aa [炭酸脱水酵素](SITEと近傍aaは1camより) | (略 → オリジナルPDB) | |
| 1cam/L+S+近傍aa(1)| (2) [炭酸脱水酵素](1ca2とも比較) | (略 → オリジナルPDB) | |
| 4cd2/L+S (1e26と同配列;他に1daj,3cd2など) | (略 → オリジナルPDB) | |
| 1cef/L+S (1cegと同配列) | (略 → オリジナルPDB) | |
| 1ceg/L+S (1cefと同配列) | (略 → オリジナルPDB) | |
| 1cgp/L+S [cAMP] (A鎖19残基・B鎖1残基) | 1cgpのChain A・D $ #0 | |
| 2cgr/L+S (L鎖5残基・H鎖8残基) | (略 → オリジナルPDB) | |
| 1cgv/L(MB1)+S [マルトース] #1 | (略 → オリジナルPDB) |
(Protein選択) |
| 1cgv/L(MB2)+S [マルトース] |
| 1cgv/L(MB3)+S [マルトース] #1 |
| 6cha/CAT [キモトリプシン] (CAT = His57 + Asp102 + Ser195)#8 | 6chaのChain A #0 | |
| 6cha/L(SBA)+S #0 不要結合削除 | |
| 1ckp/L+S (1aq1と類似配列) | 1ckp * | |
| 3cln/L+S(EF1,EF2) * ## | 3cln * [11-406 / カルモジュリン / EF-hand] | EF1 | EF2 |
| 3cln/L+S(EF3,EF4) * ## | EF3 | EF4 |
| 1cqe/L(FLP)+S | 1cqeのChain A | |
| 1crm/L+S [Hg] | (略 → オリジナルPDB) | |
| 4csm/L+S | 4csmのChain A | |
| 1cwh/L(all)+S (1cwkと同配列;他に1cwa,1cwb,1cwc,1cwf,1cwk,1cwl,1cwm) | (略 → オリジナルPDB) | |
| 1cwk/L(all)+S (1cwhと同配列) | (略 → オリジナルPDB) | |
| 1cxf/L(ACX)+S [α-シクロデキストリン] (1cxiに別Ligandで同配列あり) | (略 → オリジナルPDB) |
(Protein選択) |
| 1cxf/L(MAL_687)+S [マルトース] (1cxiと同配列;他に=1cxe,1cxh)#1 |
| 1cxi/L(MAL_687)+S [マルトース] (1cxfと同配列;他に=1cxe,1cxh)#1 | (略 → オリジナルPDB) | |
| 1cyn/L+S | (略 → オリジナルPDB) | |
| 3dap/L(N2)+S ## | (略 → オリジナルPDB) |
(Protein選択) |
| 3dap/L(INH)+S |
| 1dbp/L+S [リボース] (同配列=2dri,1drj,1drk) | (略 → オリジナルPDB) | |
| 3dfr/L(NDP)+S * (SITEにMTX含む)#0,## | (略 → オリジナルPDB) | SITE全体 | MTX |
| 3dfr/L(MTX)+S * (SITEにNDP含む)#0,## | SITE全体 | NDP |
| 1dgd/L+S (同配列=1dge)#0,1 | (略 → オリジナルPDB) | |
| 1dil/L(EQP)+S (Sは下と同/2simと同配列;他に1dim) | (略 → オリジナルPDB) |
(Protein選択) |
| 1dil/L(AXP)+S (Sは上と同/2simと同配列;他に1dim) |
| 1dra/L(APT)+S * (同配列=1dhj,1drb・3drcなど) | 1draのChain A * | |
| 1dtp/L(APU)+S ## | (略 → オリジナルPDB) | |
| 1dyr/L(TOP)+S | (略 → オリジナルPDB) | |
| 1dz8/HEM近傍 *| 同(Iヘリックス含む) * [P450] | 1dz8のChain A * #0(PDB) | |
| 1dze/L(RET)+S | 1dze * | |
| 1dze/L(L3P)+S * #1 | |
| 1dzm/L+S<A>| <B> #5 | 1dzmのChain B * #0(PDB) | A | B |
| 1e00/L+S<A>| <B> #0,5 | (略 → オリジナルPDB) | |
| 1e02/L+S<A> [ウンデカナール]| <B> #0,5 | (略 → オリジナルPDB) | |
| 1e0p/L+S | 1e0pのChain A | |
| 1e26/L+S (4cd2と同配列;他に1daj,3cd2など) | (略 → オリジナルPDB) | |
| 1e4y/L+S ## | 1e4yのChain A | |
| 1e71/L(ASC)+S [アスコルビン酸] | (略 → オリジナルPDB) | |
| 1e71/L(ASC+2SO4)+S | |
| 1e9x/L(HEM)+S * | 1e9x [P450] | |
| 1e9x/L(PIM)+S | |
| 1ea1/L(HEM)+S * | 1ea1 * [P450] | |
| 1ea1/L(TPF)+S * | |
◎参考教材2 1e9x,1ea1の各L(HEM)+S+{PIM or TPF}重ね合わせデータ
…Protein選択による表示変更無効,{PIM or TPF}のSITE略 | (上の1e9x,1ea1で閲覧) | 1e9x: S | HEM ‖ PIM
1ea1: S | HEM ‖ TPF |
| 1ea6/L(AC3)+S * [ADP] | (略 → オリジナルPDB) | |
| 1eb2/L(BPO)+S * | (略 → オリジナルPDB) | |
| 3erd/〈L+S〉 [エストロゲン受容体] #2' | 3erdのChain A | |
| 1ere/〈L+S〉 [17β-エストラジオール,エストロゲン受容体] #2 | 1ereのChain A * | |
◎参考教材3 1ere/L+S(17β-estradiol+DES〈重ね合わせ〉)
…1ereと3erdのsite部分を重ね合わせ,3erdのprotein削除
※参考:estradiolとDESのみの重ね合わせデータ → エストラジオールとDES) | − | Protein
17β-estradiol | DES |
◎参考教材4 1ere,1err,3erd,3ertの各L+S重ね合わせデータ → 別版
…Protein選択による表示変更無効 | − | 1ere: L | S ‖ L+S
1err: L | S ‖ L+S
3erd: L | S ‖ L+S
3ert: L | S ‖ L+S |
| 1eri/SITE(HRS+HYD+DNA) *$ #0 | 1eri *$ #0 | HRS | HYD | DNA |
| 1err/〈L+S〉 [エストロゲン受容体] #2' | 1errのChain A * | |
| 3ert/〈L+S〉 [エストロゲン受容体] #2' | 3ert | |
| 1eta/L+S (1thaと類似Sequence)#2 | 1etaのChain 1 | |
| 1eul/L+S<1>| <2> [Caポンプ] | 1eul | 1 | 2 |
| 1eve/L+S+CAT [アルツハイマー病治療薬ドネペジル(アリセプト)] (文献15 口絵1およびp.202図11・14のSITEとcatalyc triad) | 1eve |
(CATは200,327,440) |
| 1fbf/L(AHM)+S (1fbc〜1fbhと同配列) | (略 → オリジナルPDB) | |
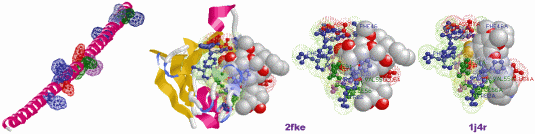
| 2fke/L+S [FK506,タクロリムス] (1j4rと同配列;他に1fkdなど) | 2fke
同配列α-helix → SITE選択 | |
| 1fld/L+S (1h61,1ofvと同Ligand) | (略 → オリジナルPDB) | |
| 1fnc/L(FAD)+S (1fndと同配列,1frnと類似配列)## | (略 → オリジナルPDB) | |
| 1fpb/L(F2A)+S (1fbfと同配列;他に1fbh) | (略 → オリジナルPDB) | |
| 1frn/L(FAD)+S (1fnc,1fndと類似配列) | (略 → オリジナルPDB) | |
| 1frp/L(AM1)+S [AMP] (1fpiと同配列) | 1frpのChain A | |
| 1frp/L(AC1)+S [FDP] (1fpiと同配列)#1 | |
| 3gbp/L+S [グルコース] (Ligand:glucose → 1gcaと比較) | (略 → オリジナルPDB) | |
| 1gca/L+S [ガラクトース] (Ligand:galactose → 3gbpと比較) | (略 → オリジナルPDB) | |
| 3gct/CAT+Chain B [キモトリプシン] (CAT = His57 + Asp102 + Ser195)#8 | 3gct | CAT | Chain B |
| 1gjm/L(HEM)+S * #0 | 1gjm | |
| 1gjm/L(FEM)+S | |
| 1gkz/L+S * [ADP] | (略 → オリジナルPDB) | |
| 1gm6/L+S * #0,1 | 1gm6 * #0 | |
| 1gm7/L+S * [ペニシリンG] | (略 → オリジナルPDB) | |
| 1gni/L(1001)+S * [オレイン酸] (OLAが7分子でそれぞれ異なるSITE) | (略 → オリジナルPDB) | |
| 1gp6/L(DH2)+S [ジヒドロケルセチン] | 1gp6 | L+S| DH2| QUE |
| 1gp6/L(QUE)+S [ケルセチン] | L+S| QUE| MES+DH2 |
| 1gqw/L(TAU)+S [タウリン] | 1gqwのChain A | |
| 1gqw/L(AKG)+S | |
| 1gvr/L+S(TNL) * #0 | 1gvr * #0 | |
| 1gwq/L(ZWB)+S * (1gwrと一部類似配列)#0 | 1gwqのChain B・D * #0 | |
| 1gwr/L+S [17β-エストラジオール] (1gwqと一部類似配列)#0 | (略 → オリジナルPDB) | |
| 1gy3/L+S [ATP] (1aq1,1ckpと一部類似配列) | (略 → オリジナルPDB) | |
| 1h11/L(FCT)+S * #0,## | 1h11 * #0,## | |
| 1h4s/L(PSD)+S * (1h4qと一部類似配列)#0 | 1h4sのChain A * #0 | |
| 1h61/L(FMN)+S * (1fld,1ofvと同Ligand) | (略 → オリジナルPDB) | |
| 1h61/L(PDN)+S * | |
| 1h7x/L(5MU)+S [抗がん剤5-FU] | 1h7xのChain A | |
| 1h9g/L(COA)+S * | (略 → オリジナルPDB) |
(Protein+Water選択) |
| 1h9g/L(MYR)+S * |
| 1h9o/L+S * #1,##(A鎖:5残基・B鎖:1残基) | 1h9o * #0 | |
| 1hbp/L+S [レチノール] (1brpと同配列) | 1hbp * | |
| 1hcq/DNA+near amino acids $ [エストロゲン受容体] ##(作者選択) | 1hcqのChain A・B・C・D $ | DNA | a.a. | All |
| 1hcq/Zinc Finger | z.f. ‖ z.f.+上記All |
| 1hd2/L+S #1 | (略 → オリジナルPDB) | |
| 1hdq/L+S * #0 | 1hdq #0 | |
| 1he1/L(AC1)+S * [GDP] (1he8と類似配列,1qf4と同Ligand) | (略 → オリジナルPDB) | |
| 1he4/L(AC1)+S * [NADP] (1he3,1he5のL(NAP)+Sと同・類似配列) | (略 → オリジナルPDB) | |
| 1he8/L+S * #0,1(1he1のL(AC1)+Sと類似配列) | 1he8のChain B * #0,1 | |
| 1het/L(NAA)+S * [NAD] #0 | (略 → オリジナルPDB) | |
| 1hfs/L(L04)+S [ポテンシャル表示不可] | 1hfs | |
◎参考教材5 1hho(O2含む),4hhbの各L(HEM)+近接His重ね合わせデータ → 別版
…SITE選択欄の指定,右下欄のProtein選択・Ligand選択,amino表示は無効
1hho・chain AのHEM+O2+近接His …[X]
4hhb・chain AのHEM+近接His …[Y] | 1hho(oxyhaemoglobin)のchain A
4hhb(deoxyhaemoglobin)のchain A | ([X][Y]と左隣欄の各Chain Aについて)
1hho: HEM近接His選択
4hhb: HEM近接His選択 |
| 1hii/L(S1P)+S (A鎖:3残基・B鎖:6残基;同配列=1hih)#0 | 1hii #0 | |
| 1hiy/L(AC1)+S | (略 → オリジナルPDB) |
(Protein+Water選択) |
| 1hiy/L(AC2)+S * #1 |
| 1hiy/L(AC3)+S |
| 1hj5/L(C1A)+S | (略 → オリジナルPDB) | |
| 1hj5/L(D1A)+S * #0 | |
| 1hj8/L(BM1)+S 〈H原子含む〉 #1,## | (略 → オリジナルPDB) | |
| 1hld/L(NDA)+S [NAD] (1hetのL(NAA)+Sと類似配列) | (略 → オリジナルPDB) | |
| 1hld/L(PFB)+S (LigandにZN・NAD含む) | |
| 1hpk/L+S (同配列=1hpj) | (略 → オリジナルPDB) | |
| 1hug/L+S [Au] | (略 → オリジナルPDB) | |
| 1iei/L(ZES)+S (SITE記載なし;1us0と同配列選択) | 1iei | |
| 1ing/L+S (同配列=1inh) | 1ingのChain A * | |
| 1inh/L+S (同配列=1ing) | 1inhのChain A | |
| 1iny/L+S (類似配列=1ing,1inh) | 1iny | |
| 2ito/L+S | 2ito * | |
| 1iow/L+S [ADP] (同配列=1iov) | (略 → オリジナルPDB) | |
| 1iwi/L+S #3 | (略 → オリジナルPDB) | |
| 1j4r/L+S (同アミノ酸配列の2fkeのSITE情報から作成;図2)#0 | 1j4rのChain A #0 | |
| 1j51/L+S #3 | 1j51のChain A | |
| 1juy/L(GNS+IMP+ASP)+S 〈H原子含む〉 (同配列=1gin,1ksz,1nht)#1 | (略 → オリジナルPDB) | |
| 1kw8/(L+S) [BPHC] (文献10 p.852口絵3の活性中心;NOあり) | 1kw8 | |
| 1kw9/(L+S) [BPHC] (文献10 p.852口絵3の活性中心;NOなし) | 1kw9 | |
| 2ldb/L(NAD)+S | (略 → オリジナルPDB) |
(Protein選択) |
| 2ldb/L(FBP)+S #1 |
| 1lgr/L+S [AMP] #1 | (略 → オリジナルPDB) | |
| 1lgt/(L+S) [DHBD / PCB] (文献10 p.852口絵3の活性中心に準拠;NOなし,His146,His210,Glu260で1kw8・1kw9と異なる) | 1lgt | |
| 1lkd/(L+S) [DHBD / PCB] (同配列=1lgt) | 1lkd | |
| 1lld/L+S [NAD] | 1lldのChain A | |
| 1lth/L+S #0 | 1lthのChain T #0 | |
| 1ml5/anticodon-codon #0 | 1ml5のChain B・C #0 | |
| 1mms/L+S $ (A鎖:3残基・C鎖:2塩基・D鎖:2塩基)#0 | 1mmsのChain A・C $ | |
| 1mup/L+S * #4 | (略 → オリジナルPDB) | |
| 1ncx/L+S(EF1,EF2) * ##(3clnを参考にSITE抽出;1nczと同配列) | 1ncx * [EF-hand / Cd] | EF1 | EF2 |
| 1ncz/L+S(EF1,EF2) * ##(3clnを参考にSITE抽出) | 1ncz * [EF-hand / Tb] | EF1 | EF2 |
| 1nip/L(ADP+2MO4)+S (A鎖:8残基・B鎖:8残基) | (略 → オリジナルPDB) | |
| 1nnb/L+S 〈H原子含む〉 | (略 → オリジナルPDB) | |
| 1nwp/L(Cu)+S (SITE記載なし;1992年ノーベル化学賞情報から選択) | 1nwpのChain A [ブルー銅タンパク質アズリン] | |
| 1ocl/L+S #0,1 | 1oclのChain A * #1 | |
| 1ofv/L+S (1fld,1h61/FMNと同Ligand) | (略 → オリジナルPDB) | |
| 1og5/L(HEC)+S | 1og5のChain A * #0 [P450(Cyp2C9)] | |
| 1og5/L(SWF)+S * #0 | |
| 2ohx/L(DMA)+S (同配列=2oxi)#4 | (略 → オリジナルPDB) | SITE全体 | NAA |
| 2ohx/L(NAA)+S [NAD] ( 〃 ) | |
| 1ol7/L(ADP)+S #0,1 | 1ol7 * | |
| 3pcb/L(ACA)+S * (A鎖:7残基・M鎖:12残基・HOH:5) | 3pcbのChain A・M *
※3pca〜3pcnに同・類似配列等 | |
| 3pcb/S * (vestigial site;A鎖:11残基・M鎖:9残基・HOH:9;類似配列=3pcd・3pckなど) | |
◎参考教材6 1pha,1phbの各HEM+PFZ重ね合わせデータ [P450]
…1phaはcamphor 5-monoxygenase(plus-isomer),1phbは同(minus-isomer)
…Protein選択による表示変更無効 | 1pha
1phb #0 | 1pha: HEM | PFZ
1phb: HEM | PFZ |
| 2pk4/L+S [6-アミノカプロン酸] | 2pk4 * #0
同配列α-helix → SITE選択 | |
| 2prg/L+S | 2prgのChain A | |
| 1psd/L+S [NAD] | 1psdのChain A | |
| 1pth/L(COX)+S #4 | 1pth | |
| 1pth/L(PER)+S | |
| 1qf4/L+S [GDP] (1qf5と同配列,1he1と同Ligand) | (略 → オリジナルPDB) | |
| 1qkt/L+S * [エストロゲン受容体] | 1qkt * | |
| 1qku/L+S [17β-エストラジオール,エストロゲン受容体] | 1qkuのChain A | |
| 1qmq/L+S * #0 不要結合削除 | 1qmq * | |
| 1qor/L+S | (略 → オリジナルPDB) | |
| 1rlc/L+S | (略 → オリジナルPDB) | |
| 1rne/L(C60)+S | (略 → オリジナルPDB) | |
| 6rsa/L+S 〈H原子含む〉 [V] (8rsa・9rsaと同配列,別Ligand)#0 | 6rsa | |
◎参考教材7(α-helix,β-sheet構造部分の水素結合学習用)
6rsaのα-helix部分の例(Tyr25-Ser32)
6rsaのβ-sheet部分の例(Val43-Val47 & Ile81-Arg85) | − | − |
| 8rsa/L+S (6rsa・9rsaと同配列,別Ligand) | 8rsaのChain A | |
| 1rta/SITE(ACT+Ribonuclease A) *$ | 1rta $ | ACT | Ribonuclease A |
| 8ruc/L+S [ルビスコ] | 8rucのChain A | |
| 1sdy/L+S [SOD] | 1sdyのChain A | |
| 2sim/L+S (1dilと同配列;他に1dim) | (略 → オリジナルPDB) | |
| 1sln/L(INH)+S * | (略 → オリジナルPDB) | SITE全体 | INH |
| 1slt/L(NAG+GAL)+S | 1sltのChain A | |
| 1sxs/L+S (1sxzと同配列) | 1sxsのChain A | |
| 1sxz/L+S (1sxsと同配列) | 1sxzのChain A | |
| 1tco/L(FK5)+S [FK506,タクロリムス] #0(A鎖:3残基・B鎖:3残基・C鎖:12残基) | 1tco #0 | |
| 1teh/L+S | 1tehのChain A | |
| 1tha/L+S (1thc,1tyrと同配列;他に2roy,1tlm/1etaと類似Sequence) | 1thaのChain A * #0 | |
| 1thc/L(130)+S (1tha,1tyrと同配列;他に2roy,1tlm/1etaと類似Sequence) | 1thcのChain A | |
| 1thl/L(PEP)+S | (略 → オリジナルPDB) | |
| 1tyr/L+S [レチノイン酸] (1tha,1thcと同配列;他に2roy,1tlm) | (略 → オリジナルPDB) | |
| 1uom/L+S * [エストロゲン受容体] | (略 → オリジナルPDB) | |
| 1us0/L(LDT)+S ##(B配座削除) | 1us0 ## | |
| 1uv6/L(AC1)+S #0 | 1uv6のChain A-E #0 [カルバミルコリン] | |
| 1uv6/L(AC2)+S #0 | |
| 1uw6/L(AC1)+S * #0 | 1uw6のChain A-E #0 [ニコチン] | |
| 1uw6/L(AC5)+S * #0 | |
| 1vfn/L+S * | (略 → オリジナルPDB) | |
| 1xva/(L+S) (文献10 p.1069図6より作成;Act294含む) | 1xvaのChain A | |
| 1yat/L+S [FK506,タクロリムス] (2fkeと類似配列) | (略 → オリジナルPDB) | |
| 1ytn/L+S | (略 → オリジナルPDB) | |