→ Jmol版データ集 | Chime版データ集 ←
※マニュアル(Chime版;Jmolもほぼ同様),収録PDBデータの加工について
★Chime版データを利用するには分子表示用プログラムのChimeが必要です(ダウンロード方法;IE7では現在利用不可)★
★Chimeコンテンツの一部はIEでエラーになる場合がありますので,Firefoxでお試しください(京都女子大・小波先生の情報)★
[TOPIC]
本コンテンツ集が国立国会図書館のPORTAに収録(旧・Dnaviおよびデジタルアーカイブポータル)
P450部分データ集について日本コンピュータ化学会2004秋季年会(2003/10/02-03,東京)で発表
PDBデータのLigand結合部位について日本コンピュータ化学会2003秋季年会(2003/10/25-26,広島)で発表;要旨
本データ集について,SCCJ2002秋季年会(2002/11/02-03,米沢)で発表;要旨
「生命情報科学概論」(新潟薬科大学での2003年度非常勤担当)の講義ページ公開 | PSBとSOSUI・TMHMMを利用した演習
関連ページ:複合分子と分子の重ね合わせ/分子データの作成方法
PDBデータを詳細に見るために‖《バーチャル分子NEWS》発行開始
バイオ関連トピックス集へ → Web上の関連ニュース紹介|検索サイトのカテゴリや最新情報検索結果例
= その他のコンテンツ例 =
※以下の多くはChime版ですが,一部Jmol版に変更しました。
生体分子のかたちの不思議 | 生体分子の構成元素
分子と分子の相互作用(QSARと有機概念図から)/1I7Z(コカインを含むデータ例)のChain A・B
コドンと遺伝暗号表/1G59のChain A・Bなど
P450部分データ集
膜貫通タンパク質データ集 | βバレル型膜タンパク質データ集
PSBとSOSUI・TMHMMを利用した演習/1F88など
糖鎖を含むタンパク質/2MSBのChain Bなど
ノイラミニダーゼ立体構造予測データベース(理研)のデータより/AAC14419.1(理研DB)
科学未来館展示のタンパク質/1IGTなど
アプタマー/1KOCほか
ラメラリン α20-サルフェート(HIVインテグラーゼ阻害剤)/1QS4のChain A
対称構造を持つオリゴマータンパク質の例/1G31など
コレラ毒素の例/1XTC・1LTTのChain D-H
制がん剤/151Dの部分(DNAへのadriamycinのインターカレーション)と1TUP(がん抑制遺伝子p53)のDNA+Chain B
抗がん剤開発と標的タンパク質/1ISAなど
発がん性化合物の例/発がん性分子代謝物のDNA修飾など(1DXA・1AG5より)
セカンドメッセンジャー/1g9qのChain Aなど
抗生物質を含むPDBデータ/1EI2のModel 1など
HIVとエイズ/1OHRなど
レトロウイルスのキャプシド六角環構造/1U7K
「予防原則」を取り上げた本から/3ERD
ペニシリンGを含むPDBデータ例/1FXVのChain B
PCBの生分解反応(バイオレメディエーション)/1LKDなど
3D-QSARによる内分泌かく乱物質のスクリーニング/エストロゲン受容体の分子モデル例(1ere等より)
エストロゲン受容体4種のSite部分重ね合わせ(1ere,1err,3erd,3ertより)
ヘモグロビン/1HHOと4HHBのChain A
世界最小のタンパク質シニョリン/1UAOのModel〜16
水溶性ビタミンと脂溶性ビタミン/1E0P・1BU5・1BJN・1FE7のChain Aなど
風邪ウイルスに直接効く薬・プレコナリル/1C8MのMol_Id 1
緑色蛍光を発するタンパク質・GFP/1HCJのChain A
eF-siteとProModeを見るために/1AQU・1BBHのChain Aなど [2002年ノーベル化学賞情報参照!]
《 今後も追加します;今週の分子も参照 》
★本サイト開設7周年記念のエッセー風コンテンツ“DNAの脆弱性と強靭性”にも作成画像を多数掲載しています.
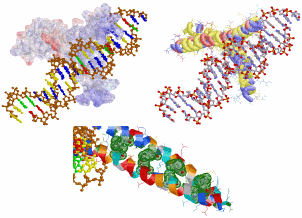
画像作成例1 PDBコード順データリストページで作成したDNA接合タンパク質(1a02のChain F・J+DNA)の表示例.
上左:タンパク質は静電ポテンシャル表示でDNAは塩基別表示,上右:タンパク質アミノ酸残基の酸性・中性〈芳香族〉・塩基性区別表示.
下:PDBのLigand結合部位ページで作成したLeucine Zipper部分(緑色のDot Surface表示)
※Leucine Zipperについては,「できるバイオインフォマティクス」p.83,「ゲノム 第2版」,p.277参照.
※JmolによるPDBデータ表示例でJmolでの1a02データ参照可能.
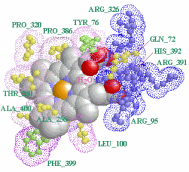
画像作成例2 PDBデータのLigand結合部位ページで作成した1ea1の例.アミノ酸は酸性・中性・塩基性区別,Dot Surfaceはlog P値順表示した場合.
※アミノ酸および特性基の親水性・疎水性参照
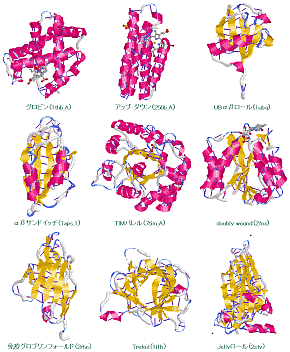
画像作成例3 スーパーフォールドの例(水素結合表示,Ligandは球棒モデル)
グロビン(1thb_A),アップ-ダウン(256b_A),UBαβロール(1ubq),αβサンドイッチ(1aps_1),TIMバレル(7tim_A) doubly wound(2fox),免疫グロブリンフォールド(2rhe),Trefoil(1i1b),Jellyロール(2stv)
※美宅成樹・榊佳之 編,「バイオインフォマティクス」,p.175,東京化学同人(2003)
※SCOP: Structural Classification of Proteins | CATH database of structural domains
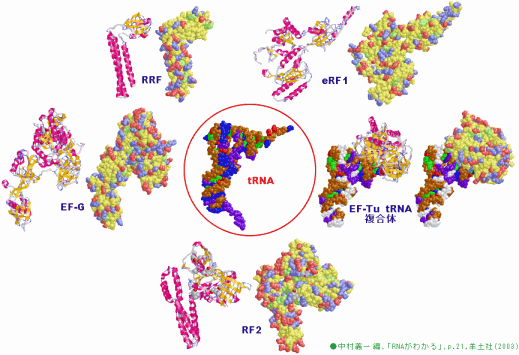
画像作成例4 PDBデータLigand結合部位ページで作成したtRNA擬態タンパク質の例(下記文献p.21図2を参考に作成;コドンと遺伝暗号表も参照)
[上]左から,RRF;(1eh1_A),eRF1(1dt9_A)
[中]左から,EF-G(1dar_A),tRNA(ここでは1ml5_BC),EF-Tu tRNA 複合体(1ttt_AD)
[下]RF2(1gqe_A;原報と分子の向きが異なる)
◎詳細:中村義一 編,「RNAがわかる」,p.21,羊土社(2003)
※Googleによる“tRNA 擬態”検索結果 | 同“分子擬態”
※分子擬態の解説例:タンパク質の生合成(福岡大学大学院理学研究科)
※図(リボンモデルと酸性・中性・塩基性アミノ酸区別表示)とPDB部分データ作成に当たっては,2003年度ゼミ生の金子祐子さんの協力を得ました.