- もうひとつの驚異の人類史 『大図鑑 コードの秘密』訳者の浜口稔さんに聞く(じんぶん堂,2021/03/24)
- 本の学校から コード読み解く秘密(朝日 鳥取版,2021/03/24)
- 「大図鑑 コードの秘密 世界に隠されたメッセージを読み解く」(明石書店) - Togetter
- 奔流する図版は誰に何を伝えるのか - Amazonカスタマーレビュー ※《注1》の有機概念図についてはこちらを参照
- 元素・分子が登場する本を読もう!(本サイト)
(本ページ作者の旅行記録から)




スペイン旅行(2016年)から
「大図鑑 コードの秘密」に出てくるキーワード関連化合物等:五十音順,最初に数字・アルファベット
初期表示は金の結晶構造(“錬金術”関連)
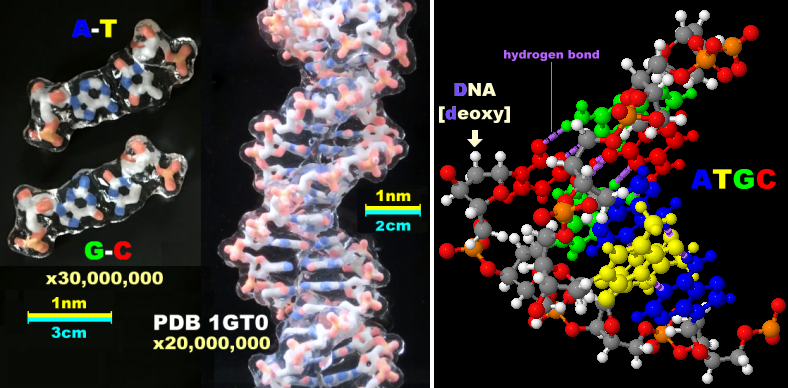

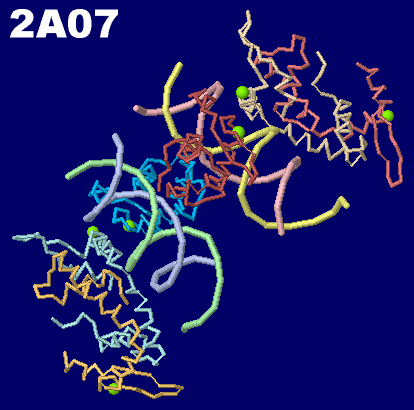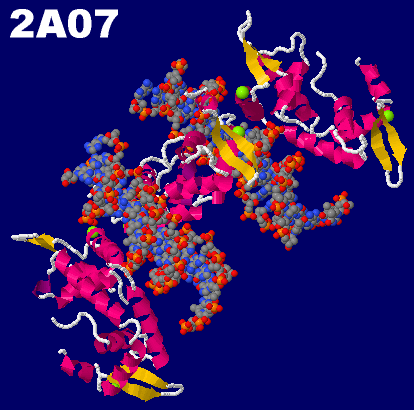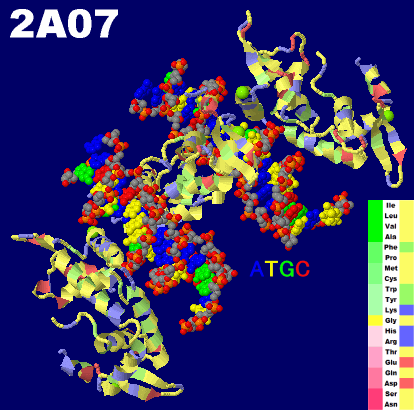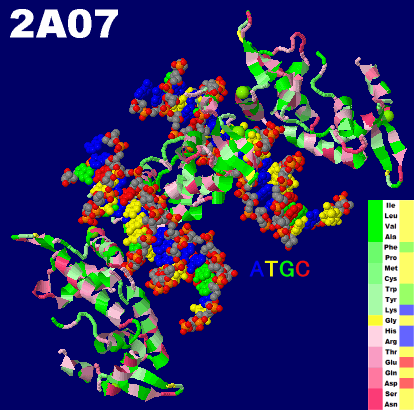
[左]DNA部分構造例とそれを構成する4種類の塩基 → DNAとRNAのいろいろな姿
[中]ダイヤモンド・金・食塩(塩化ナトリウム)などの結晶構造 → 川上モデル|分子・結晶・リガンド模型
[右]DNAに結合した言語関連遺伝子FOXP2のフォークヘッドドメインの構造2a07
-
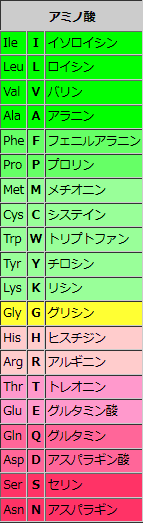
●アミノ色表示の凡例
ASP GLU CYS MET LYS ARG SER THR PHE TYR ASN GLN GLY LEU VAL ILE ALA TRP HIS PRO
●酸性・中性〈芳香族〉・塩基性アミノ酸区別表示の凡例
ASP GLU GLY ALA VAL LEU ILE CYS SER THR ASN GLN PRO MET PHE TYR TRP LYS ARG HIS
●極性〈酸性・塩基性〉・非極性(疎水性)アミノ酸区別
SER THR TYR CYS ASN GLN ASP GLU LYS ARG HIS GLY ALA VAL LEU ILE PHE PRO MET TRP
●疎水性インデックス順
ARG LYS ASN ASP GLN GLU HIS PRO TYR TRP SER THR GLY ALA MET CYS PHE LEU VAL ILE
●有機概念図I/O値順(特性基 R)
ASN SER ASP GLN GLU THR ARG HIS GLY LYS TYR TRP CYS MET PRO PHE ALA VAL LEU ILE
●等電点順
ASP GLU CYS ASN PHE GLN TYR SER MET TRP VAL GLY LEU ALA ILE THR PRO HIS LYS ARG
アミノ酸および特性基の親水性・疎水性 | Log Pをポケットに!
抗マラリア物質クラドスポリン(cladosporin)が結合した熱帯熱マラリア原虫のリシルtRNAシンターゼ4ycvのChain A・B
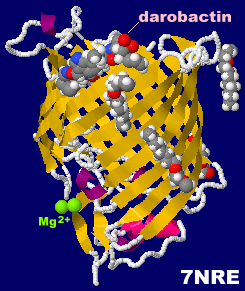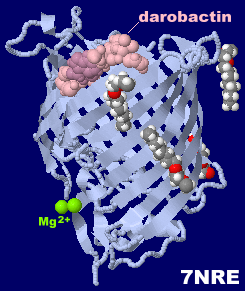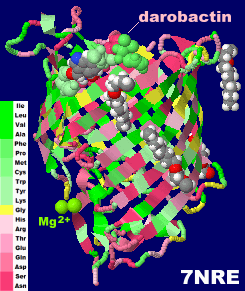
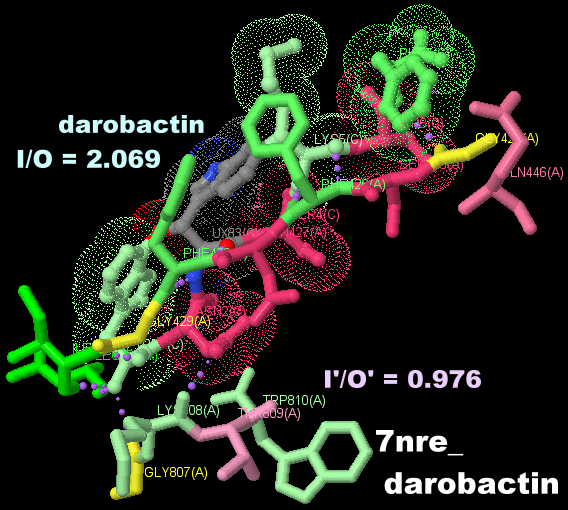
ダロバクチン(darobactin)が結合したBAM(β-barrel assembly machinery)複合体構成要素BamA 7nreとPDBsumデータ(ドット表面表示がdarobactin)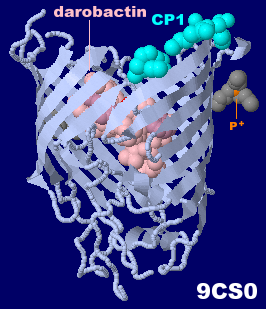
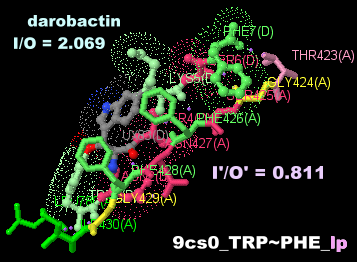
ダロバクチン(darobactin)とCP1が結合したBAM(β-barrel assembly machinery)複合体構成要素BamA 9cs0とPDBsumデータ(ドット表面表示がdarobactin)
[Fetal hemoglobin - Wikipedia]
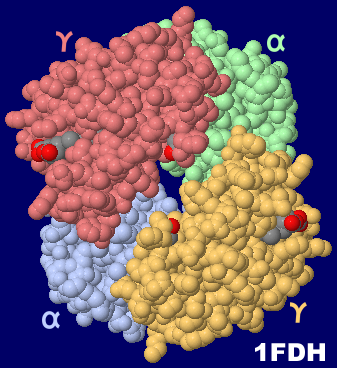

[左]胎児ヘモグロビンHbF(1fdh) → PDBj今月の分子 257: 胎児ヘモグロビン
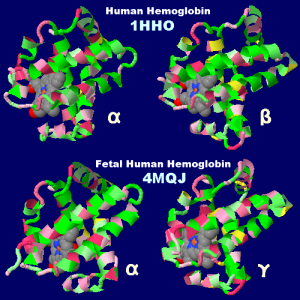
[中]成人のヘモグロビンHbA(1hho;α2β2)と胎児ヘモグロビンHbF(4mqj;α2γ2)の比較
[右]成人のヘモグロビンHbA 2hhb(α2β2)の川上モデル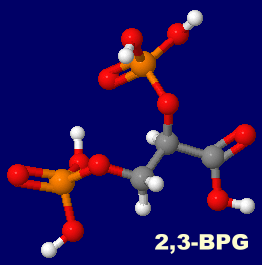
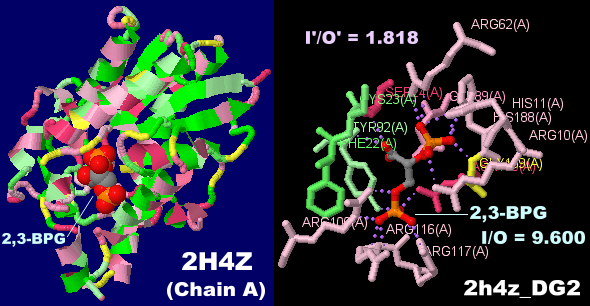
[左]2,3-ビスホスホグリセリン酸(2,3-BPG) [中・右]同分子結合ビスホスホグリセリン酸ムターゼ2h4zのChain AとPDBsumデータ
[Nicotinic acetylcholine receptor - Wikipedia,Alpha-7 nicotinic receptor - Wikipedia]
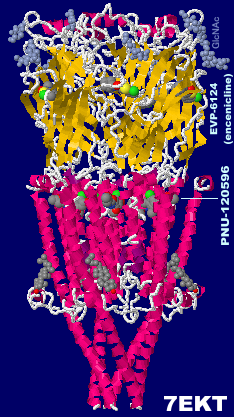
EVP-6124(encenicline;エンセニクリン)とPNU-120596が結合したヒトα7ニコチン性アセチルコリン受容体 7ekt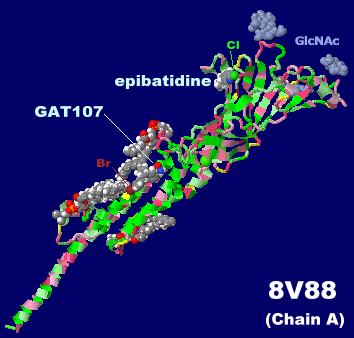
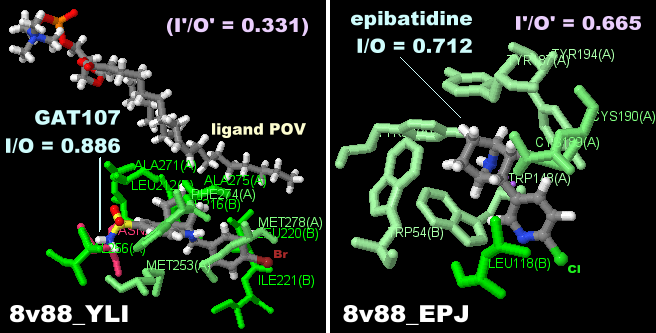
GAT107とエピバチジン(epibatidine)が結合したヒトα7ニコチン性アセチルコリン受容体 8v88のChain AとPDBsumデータ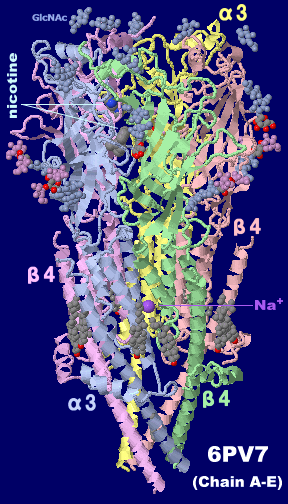
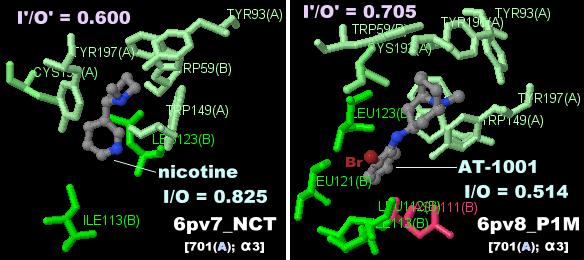
ニコチンが結合したヒトα3β4ニコチン性アセチルコリン受容体6pv7のChain A-EとPDBsumデータ(AT-1001結合の6pv8との比較)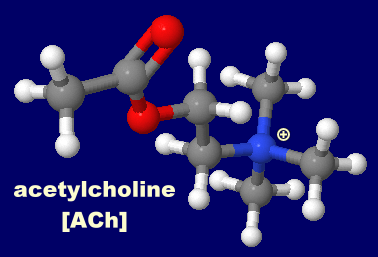
[左]アセチルコリン(acetylcholine,ACh)
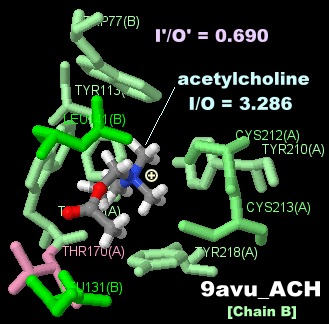
[中・右]アセチルコリンが結合したウシのアセチルコリン受容体9avuのChain B(βサブユニット)とPDBsumデータ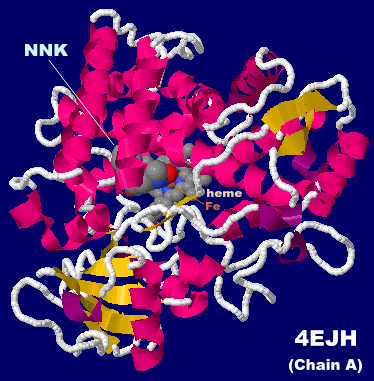
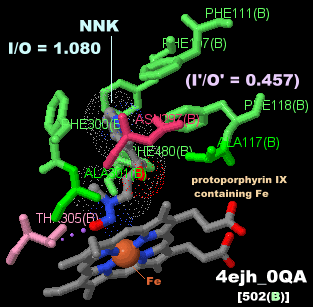
NNKが結合したP450 2A13 4ejhのChain AとPDBsumデータ

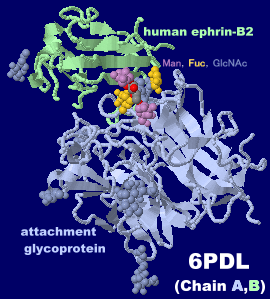
ヒトの受容体エフリンB2と結合したヘンドラウイルスG糖タンパク質6pdlのChain A・B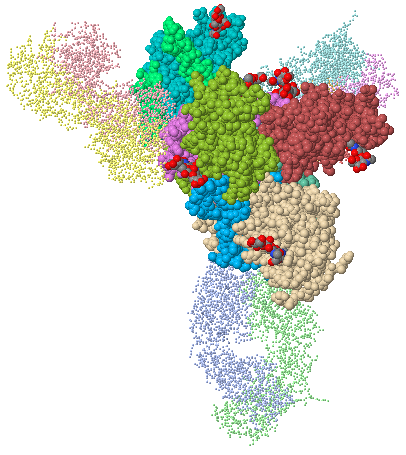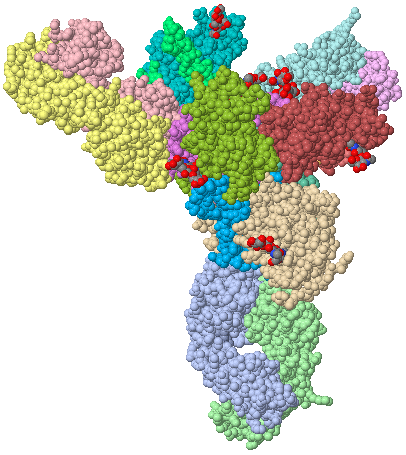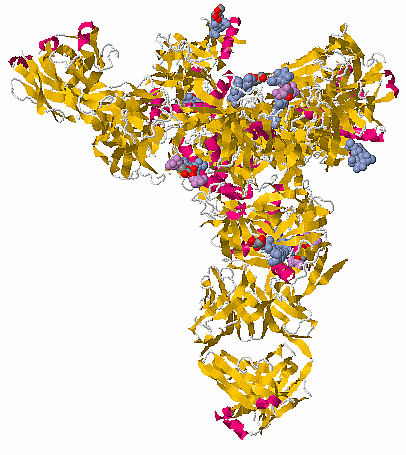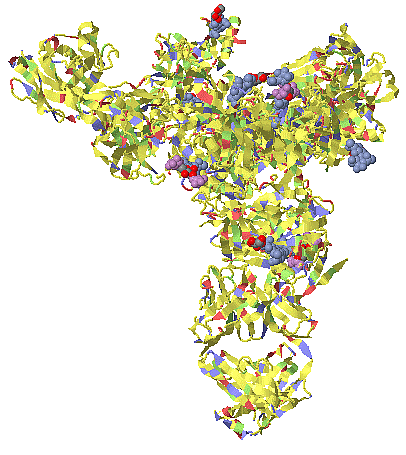
ヒト由来抗体が結合したエボラウイルス糖タンパク質3量体3csy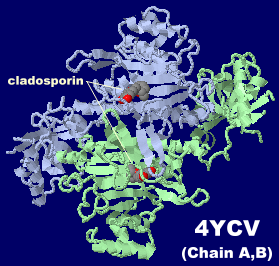
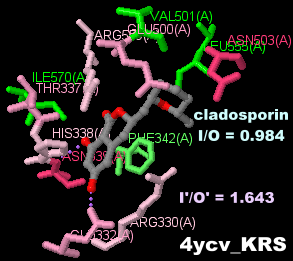
抗マラリア物質クラドスポリン(cladosporin)が結合した熱帯熱マラリア原虫のリシルtRNAシンターゼ4ycvのChain A・BとPDBsumデータ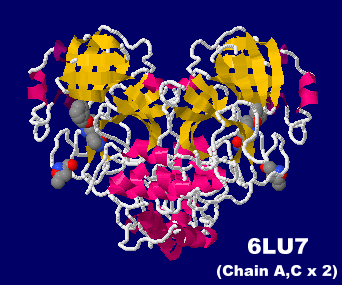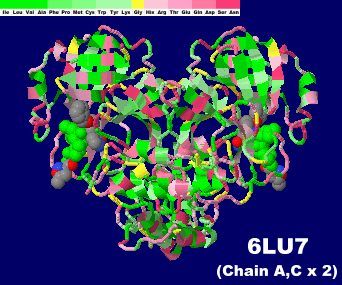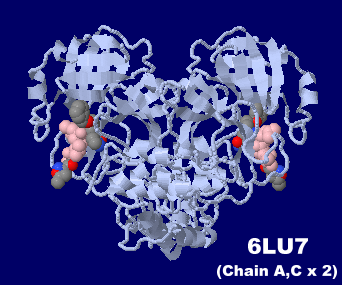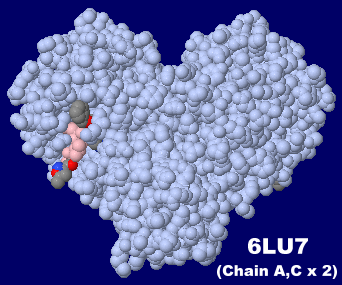
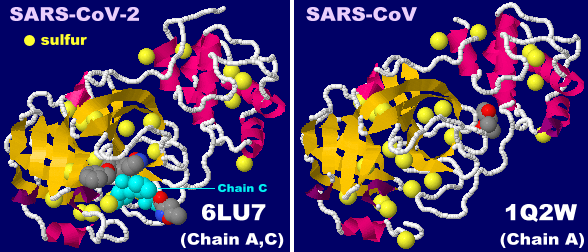
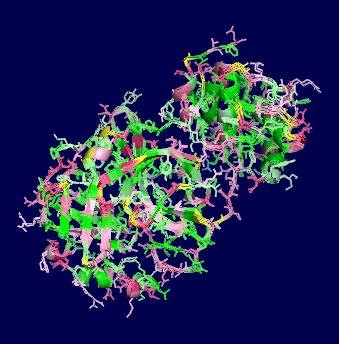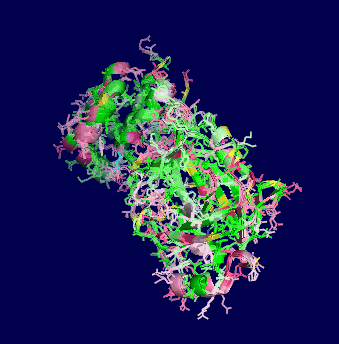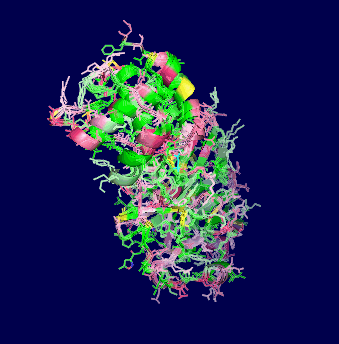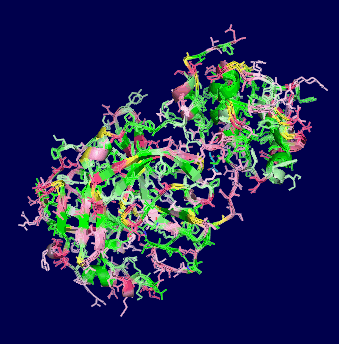
[左]新型コロナウイルスSARS-CoV-2のプロテアーゼ6lu7の4量体(Chain A・C×2)
[中・右]SARS-CoV-2のプロテアーゼ6lu7(Chain A・C)とSARSコロナウイルスのプロテアーゼ1q2wのChain Aの比較およびPyMOLによる両者の重ね合わせ画像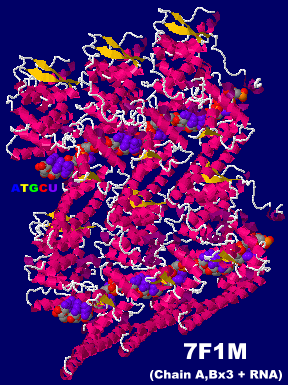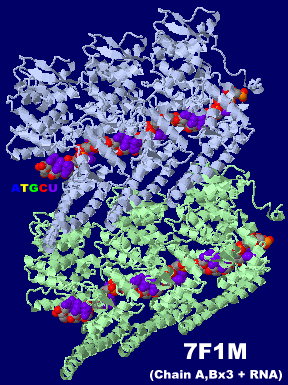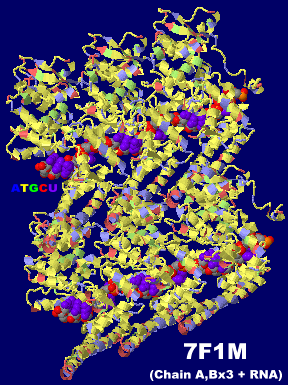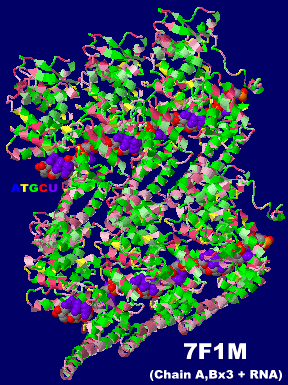
[左]マールブルグウイルス核タンパク質-RNA複合体7f1mの6量体(Chain A・B×3+RNA);らせん構造の一部
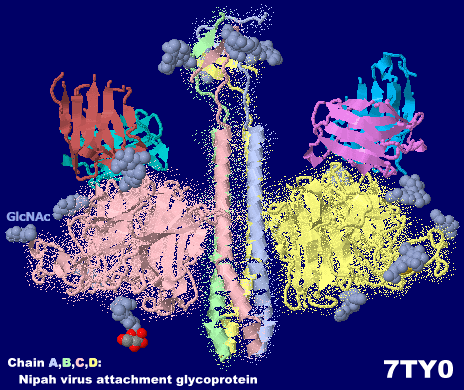
[右]中和抗体Fabフラグメントが結合したニパウイルス付着糖タンパク質7ty0(ドット表面表示のChain A-Dがニパウイルス由来)
[Nitric-oxide reductase - Wikipedia]
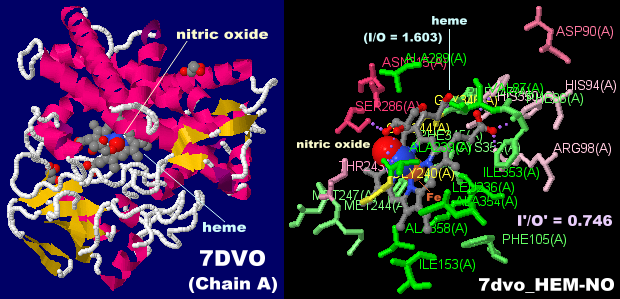
一酸化窒素が結合したP450一酸化窒素還元酵素7dvoのChain AとPDBsumデータ
[GSDMD - Wikipedia]
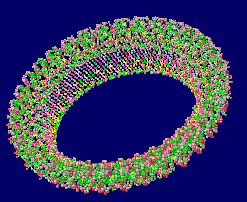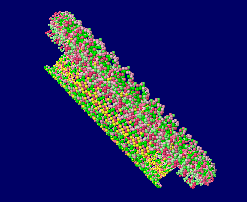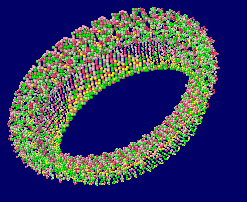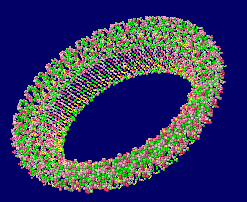
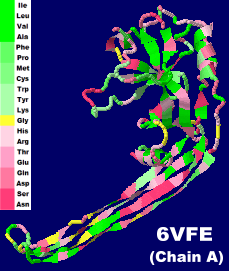
ヒトガスダーミンD 6vfe(α炭素のみ)と同Chain A全体構造
[Glucocorticoid receptor - Wikipedia,Dexamethasone - Wikipedia]
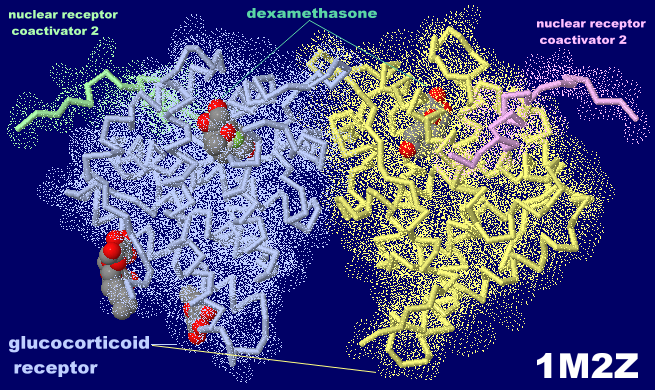
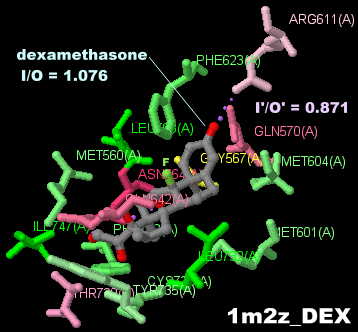
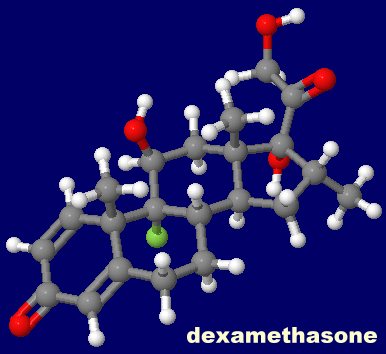
[左・中]デキサメタゾンが結合したグルココルチコイド受容体1m2zとPDBsumデータ [右]デキサメタゾン(dexamethasone)
[Protein-arginine deiminase - Wikipedia]
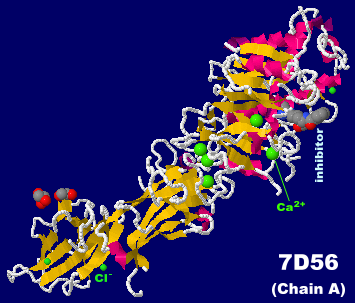
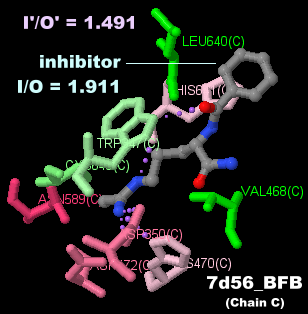
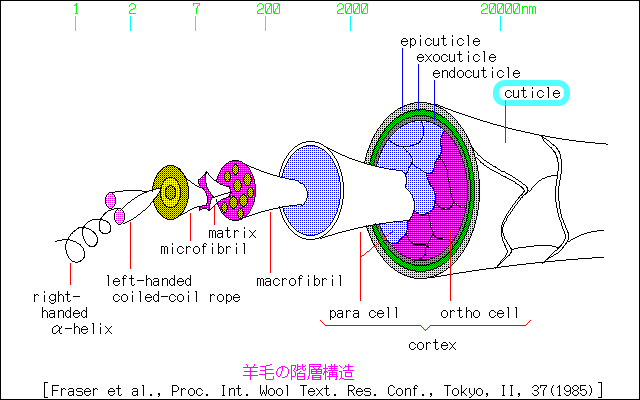
[左・中]ペプチジルアルギニンデイミナーゼ3(PAD3) 7d56のChain AとPDBsumデータ [右]参考図:羊毛のキューティクル(クチクル;水色枠)
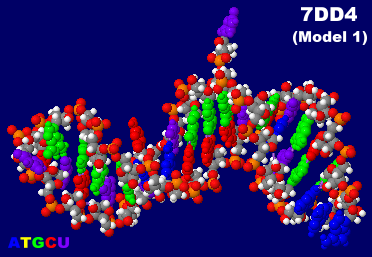
HIV-1ゲノムRNA(G1G)7dd4のModel 1



