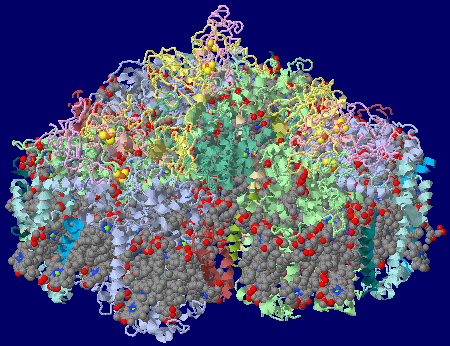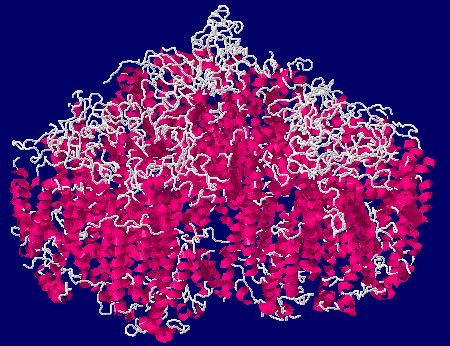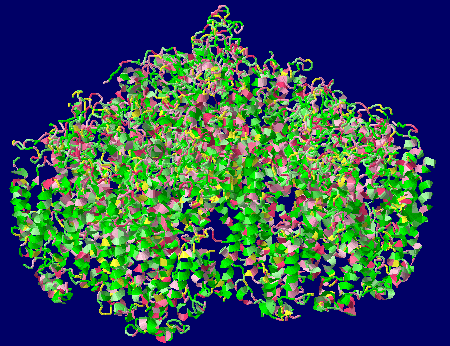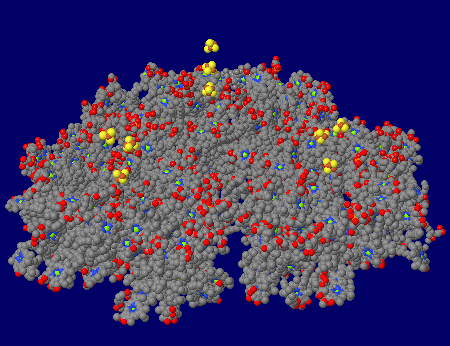
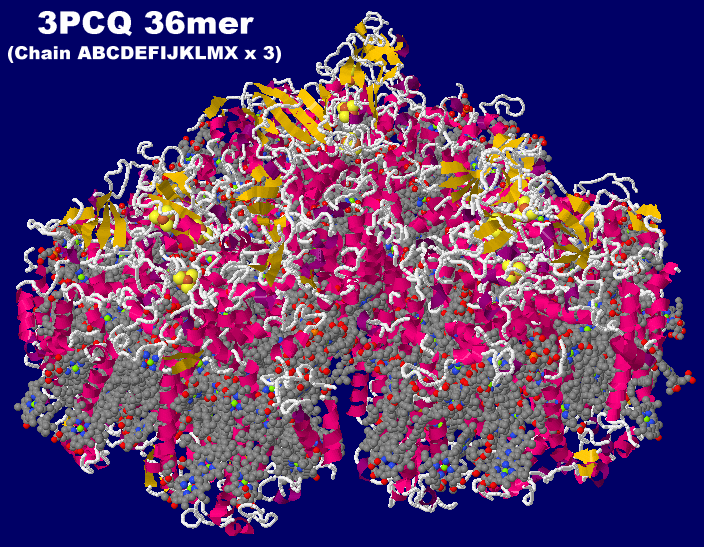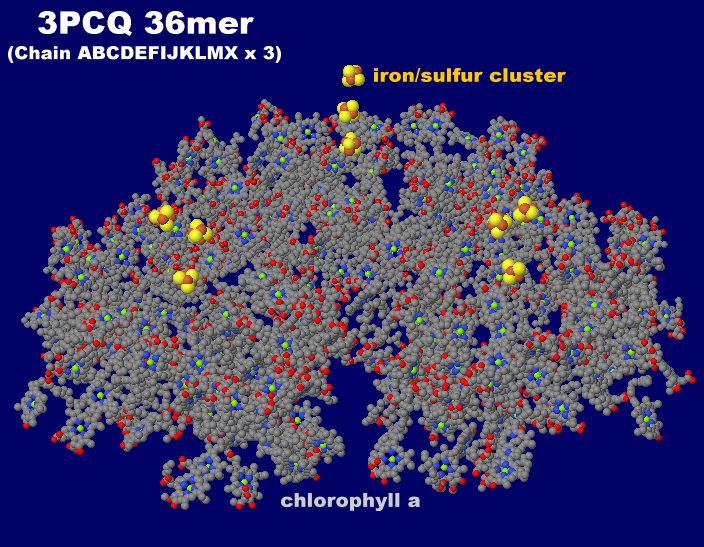
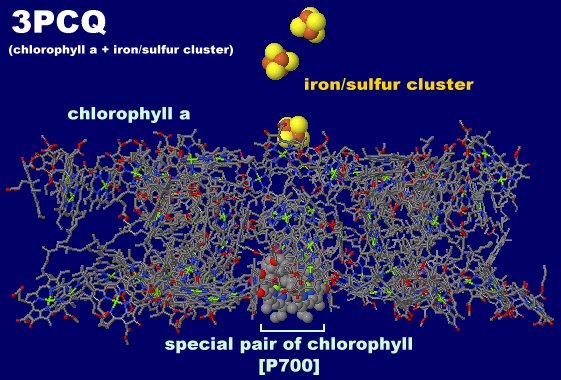
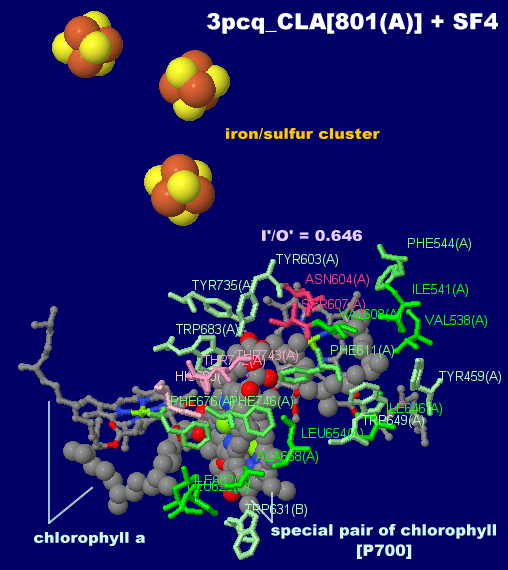
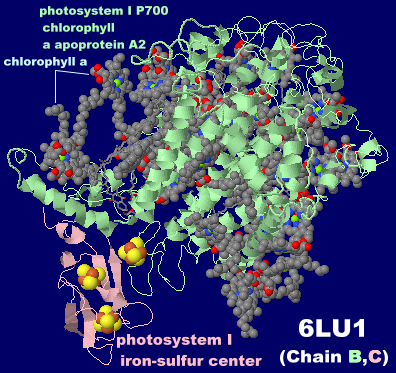
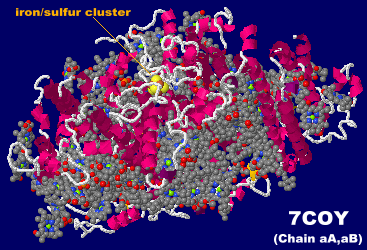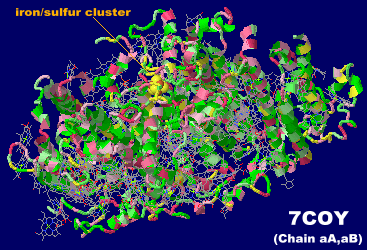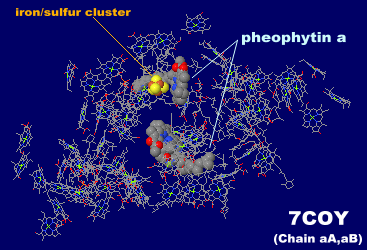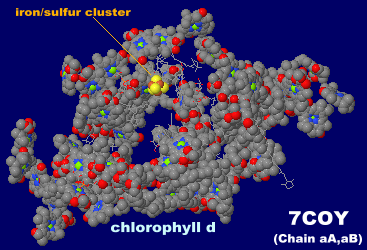
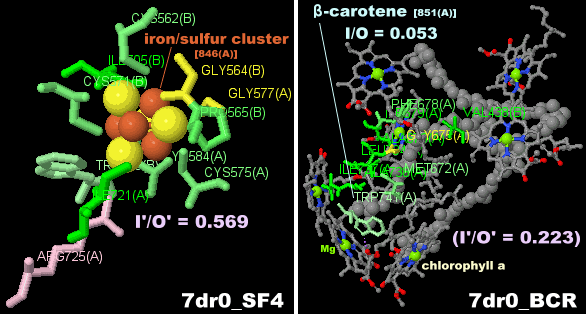
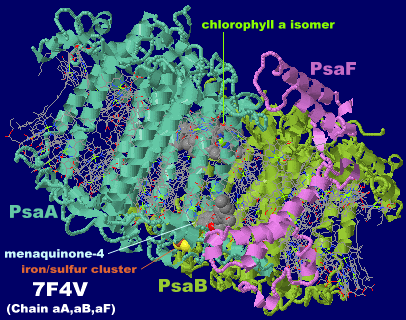
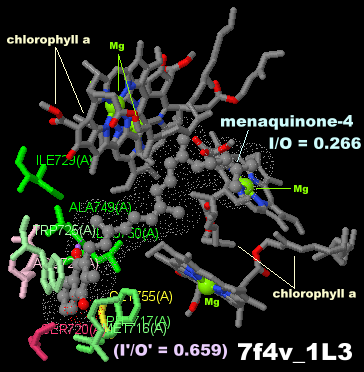
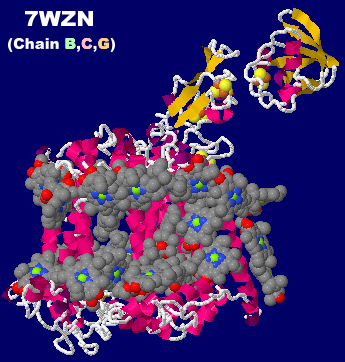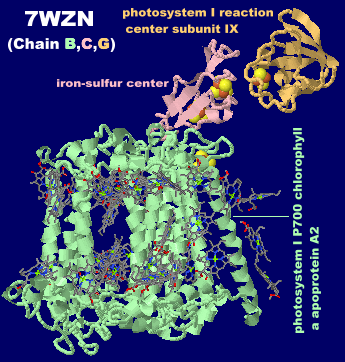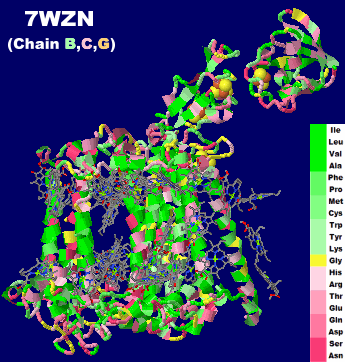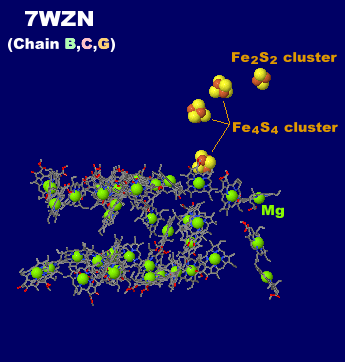
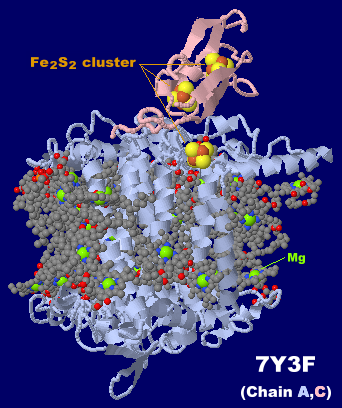
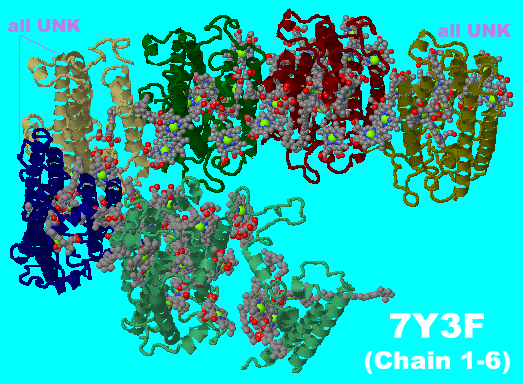
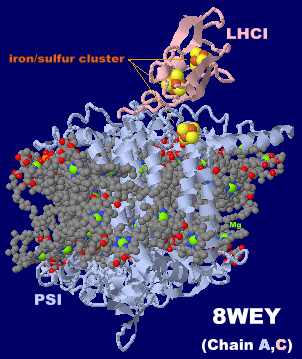
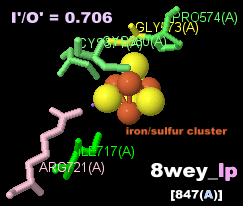
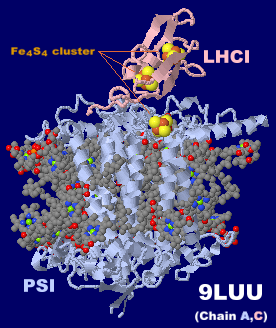
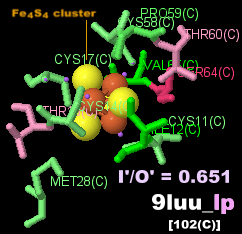
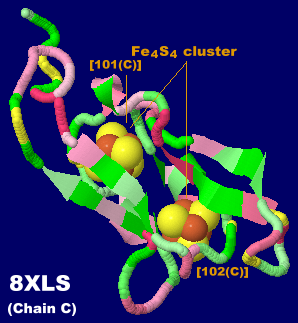
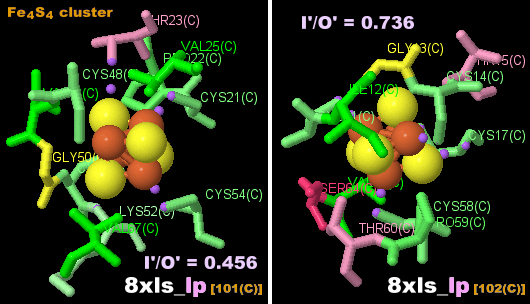
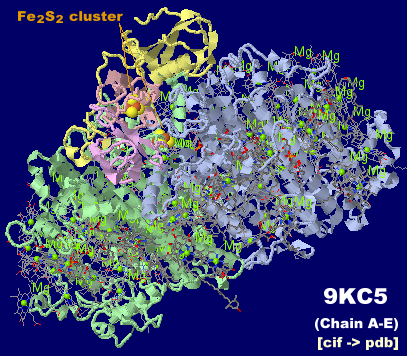
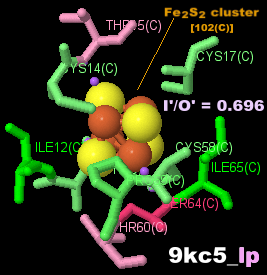
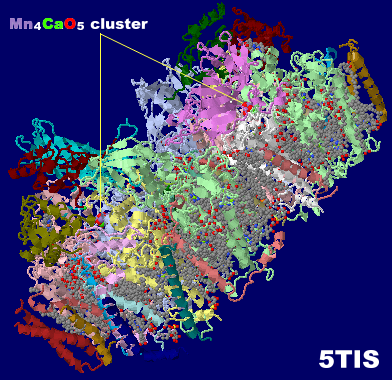
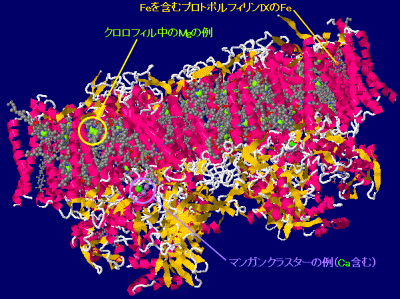
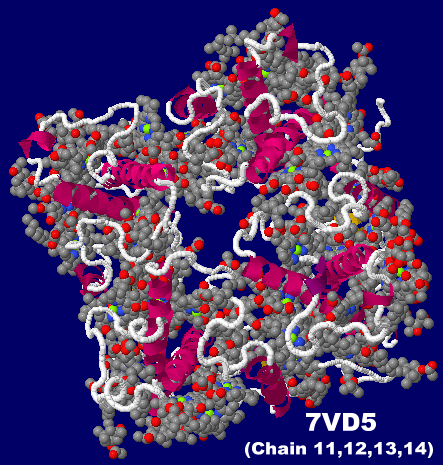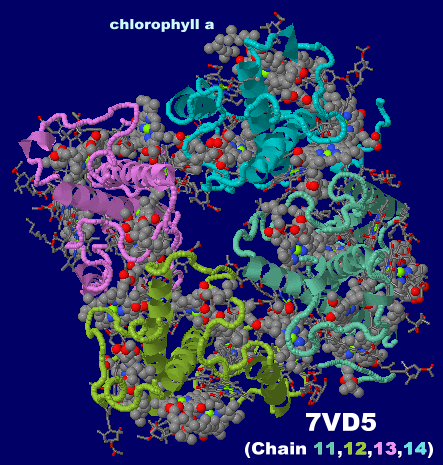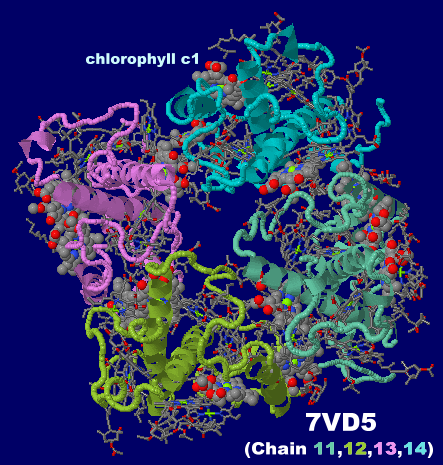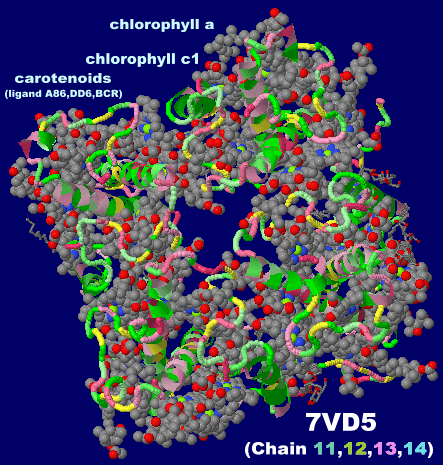
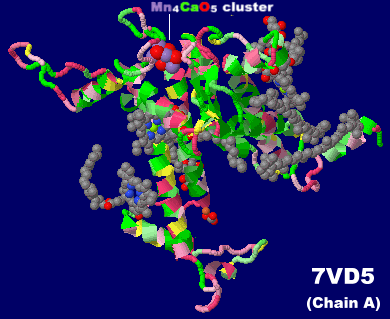
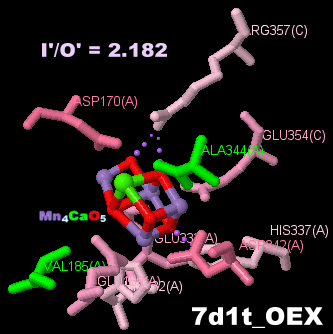
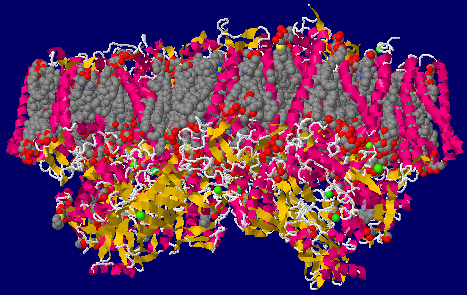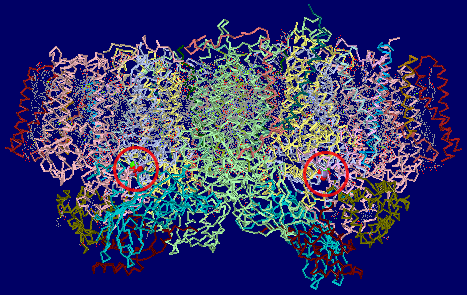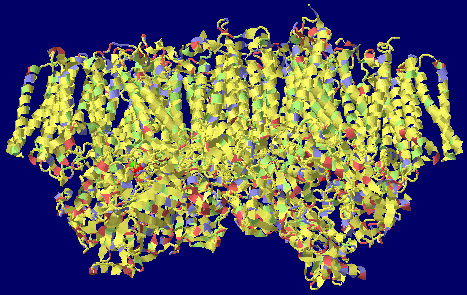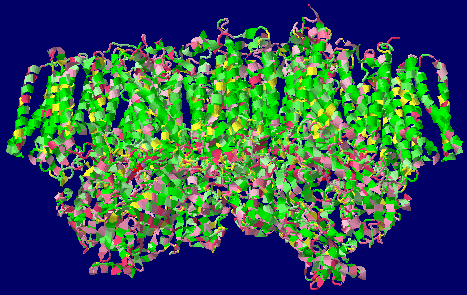
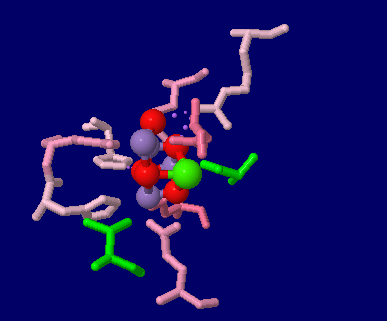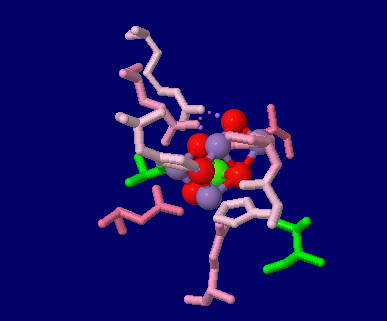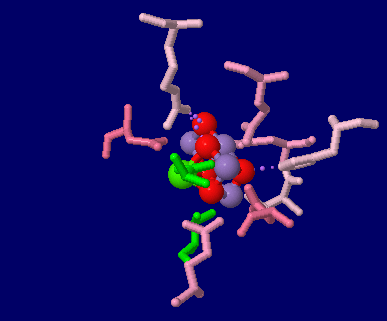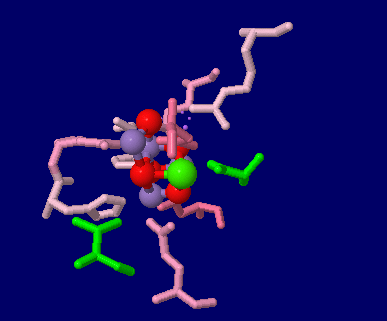
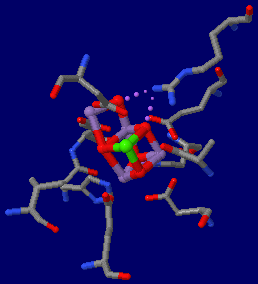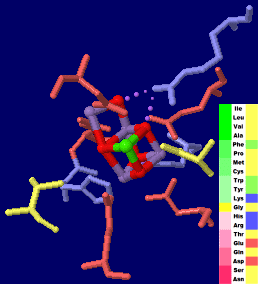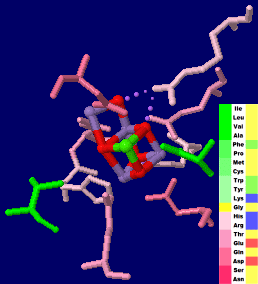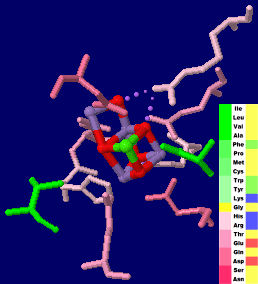
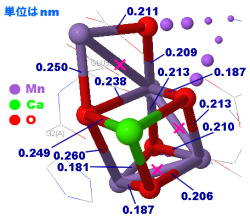
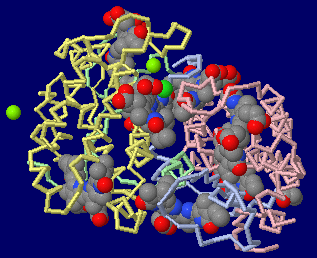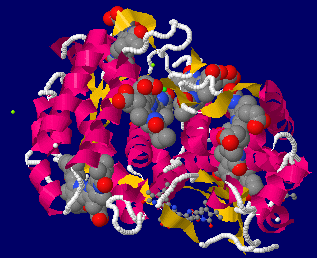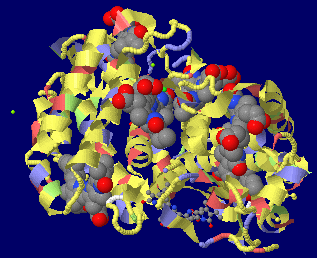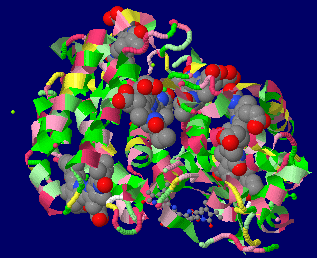
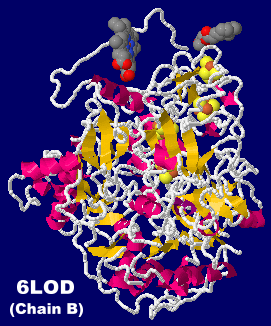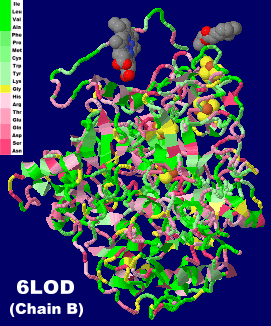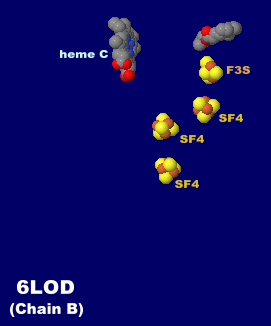
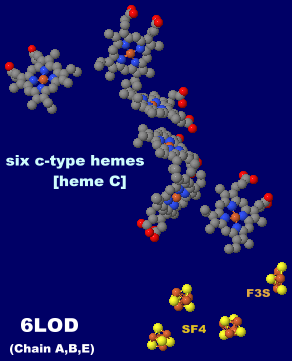
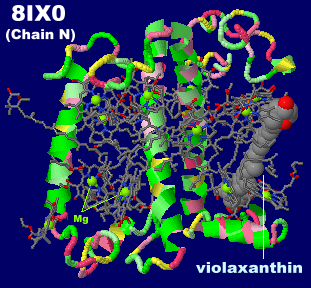
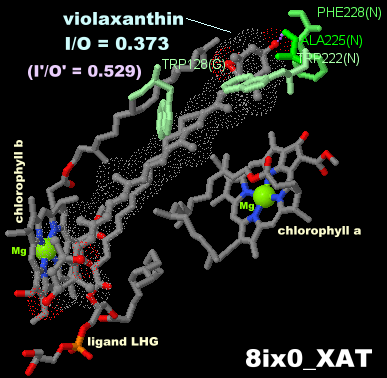
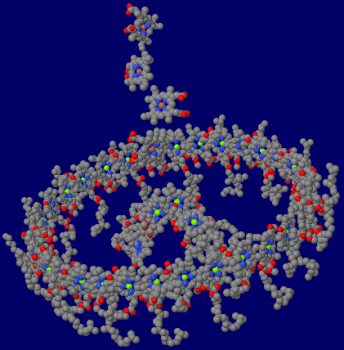
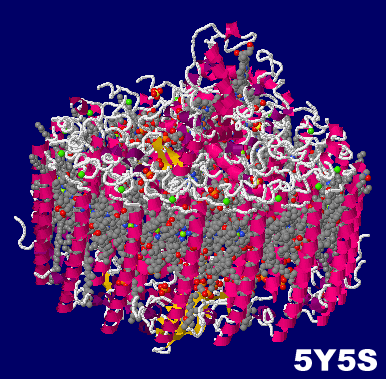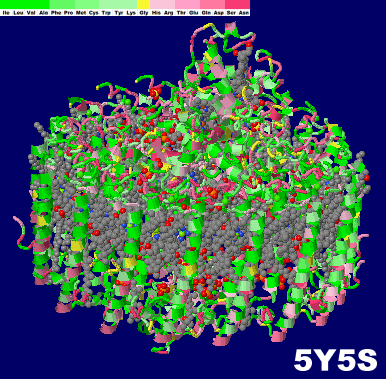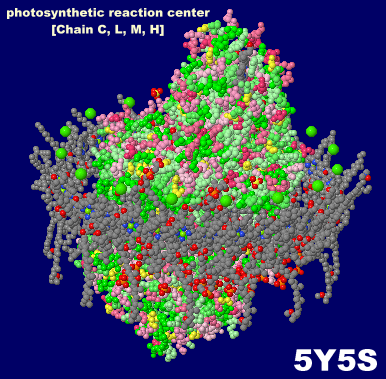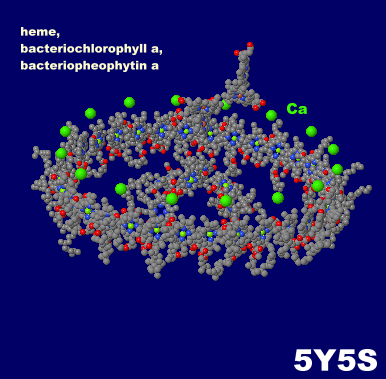
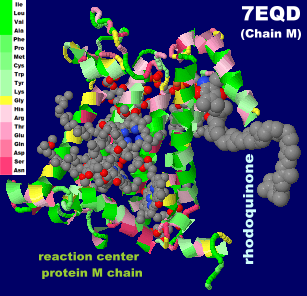
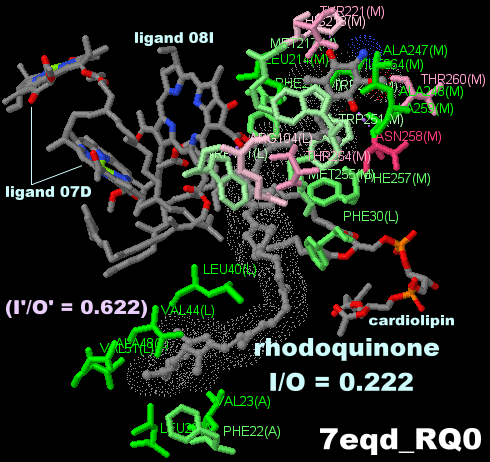
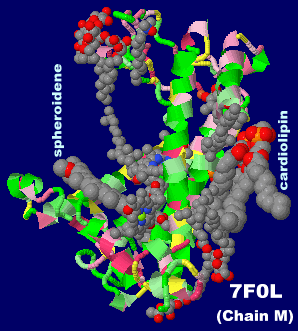
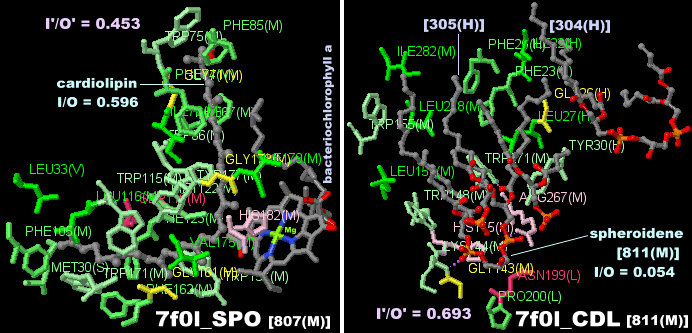
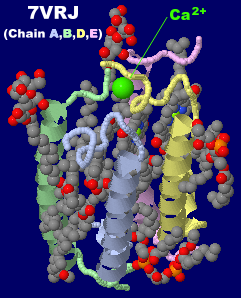
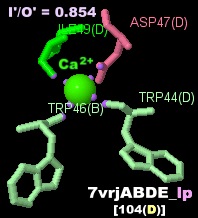
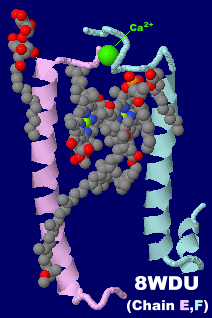
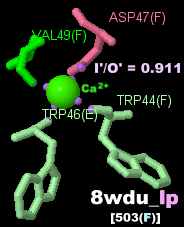
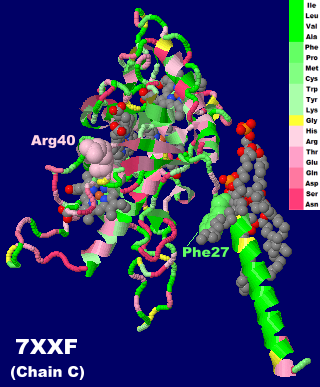
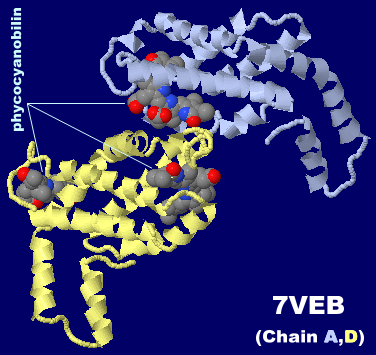
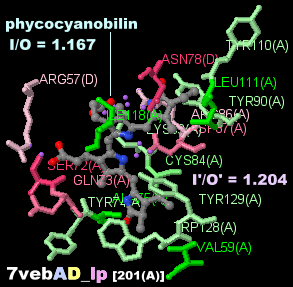
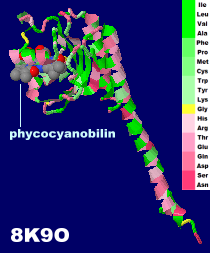
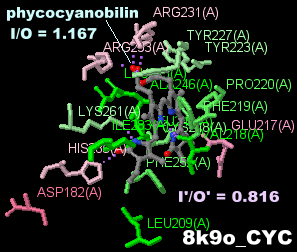
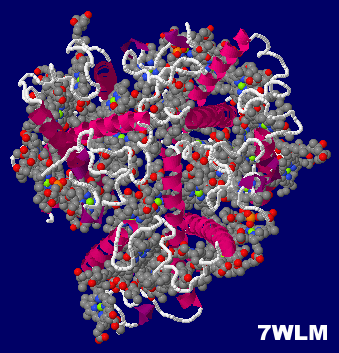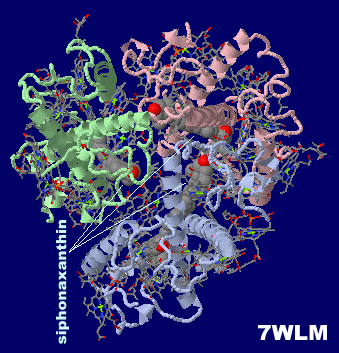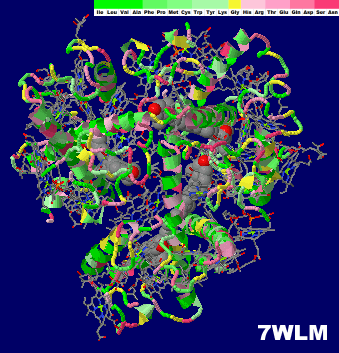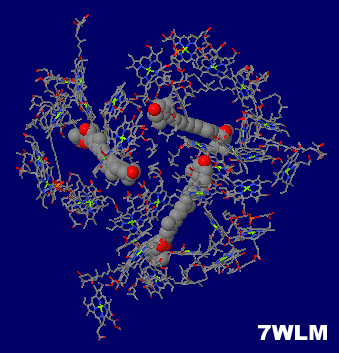
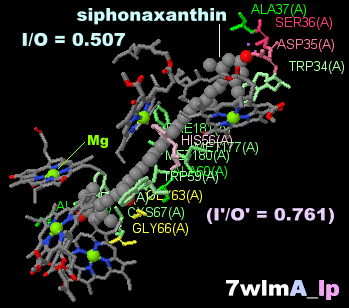
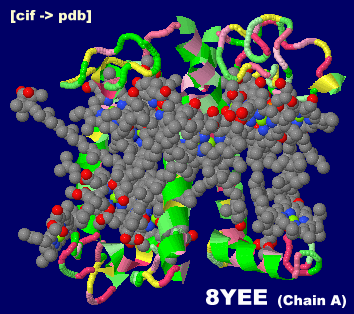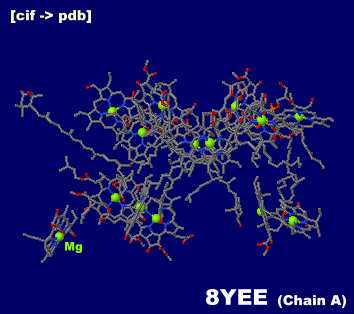
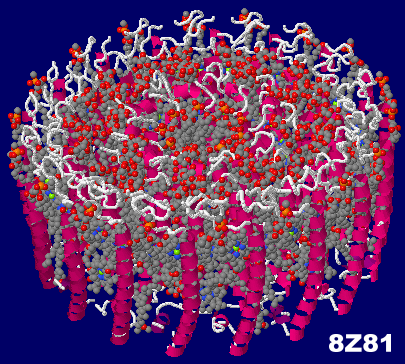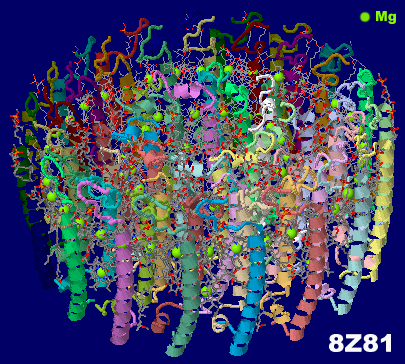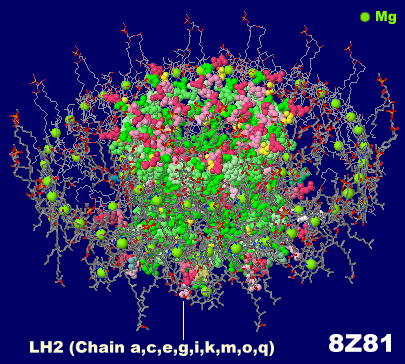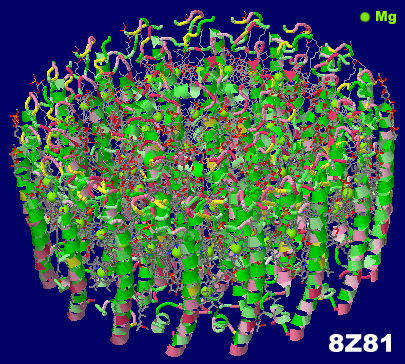
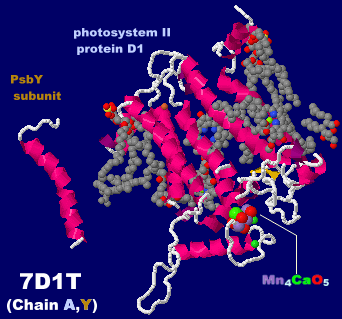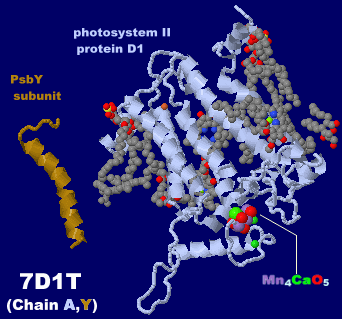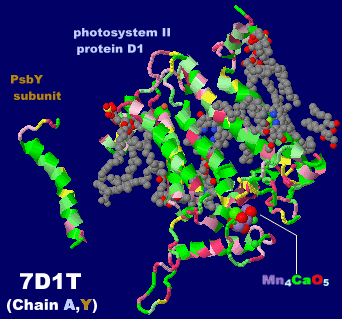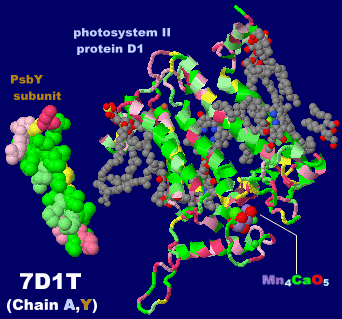
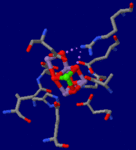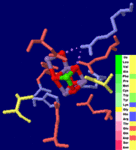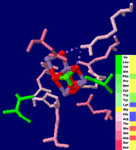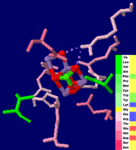
→ 光合成に関係する生体分子PDB 3ARCの部分構造例 ←
→ 生命を知り生命に学ぶ | PDBsumのLigand-SITE情報と有機性・無機性 | 金属原子を含む生体分子のSITEデータ集 ←
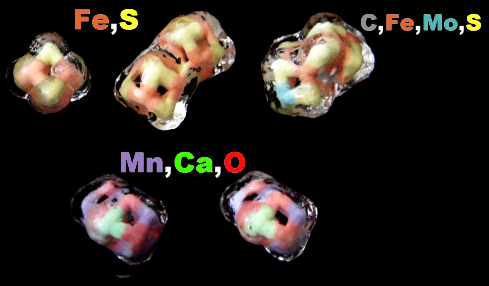
→ 鉄硫黄クラスターを含む生体分子のSITE例 | マンガンクラスターを含む生体分子のSITE例 ←
→ ミトコンドリア関連生体分子のSITE例 ←
※本ページの分子モデルはInternet Explorerで参照できます。Microsoft EdgeではInternet Explorerモードにして。
※PDBsumデータでファイル名*_lpであるものはLigPlot+ v.2.2で取得したものです。
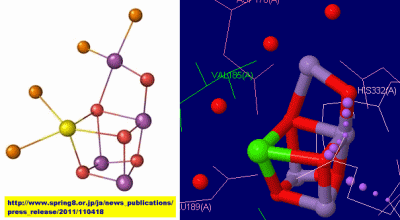
[TOPIC] 大阪市立大学と岡山大学のチームによる研究がScience誌のBreakthrough of the Year, 2011に選ばれました!!

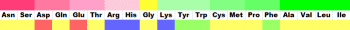
アミノ酸および特性基の親水性・疎水性 | Log Pをポケットに!
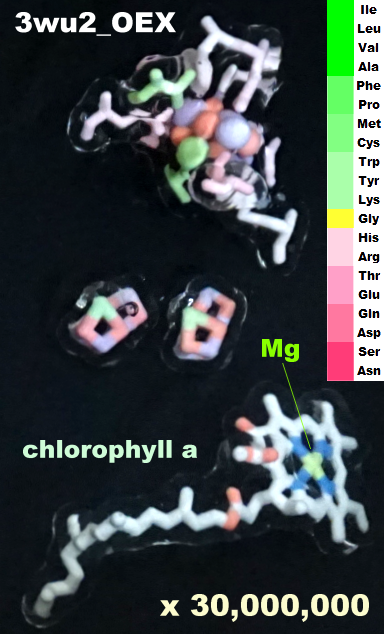
光化学系I関連 ‖ 光化学系II関連|別例(Mn4CaO5クラスター含む) ‖ 集光性タンパク質関連ほか|光合成反応中心-集光アンテナタンパク質複合体
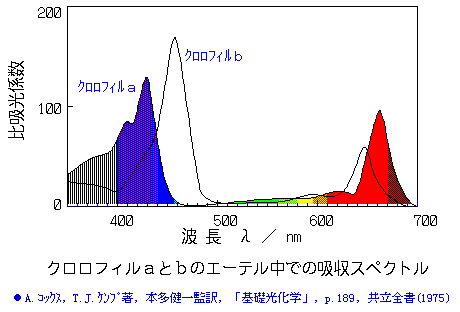
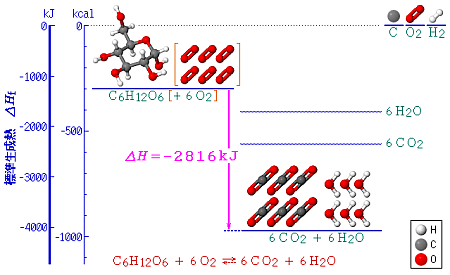
光合成の反応式(簡略式) ‖ 文献・参考ページ(ニュース例)