- Synthesis of a carbon nanobelt(Science,2017/04/14)
- Synthesis of a carbon nanobelt - 伊丹研究室 - 名古屋大学
- 名大、「カーボンナノベルト」初合成 単一構造のCNTに道(日刊工業新聞,2017/04/14)
- できた! 夢のカーボンナノベルト 60年前に存在予言(朝日,2017/04/14)
- “夢の炭素分子”「カーボンナノベルト」合成 名古屋大が世界初(ITmedia,2017/04/14)
- 「夢の筒状炭素分子」の合成に成功 名古屋大グループが世界初(マイナビニュース,2017/04/17)
- 名大、新炭素分子を合成 電子材料に期待(日経,2017/04/16)
- カーボンナノベルトの合成に成功、カーボンナノチューブ実用化への道を拓く 名古屋大学(大学ジャーナルオンライン,2017/04/22)
- カーボンナノベルト合成成功!(有機化学美術館・分館,2017/04/15)
- Winmostar: Modeling of a Carbon Nanobelt - YouTube
- フラーレン(本サイト)
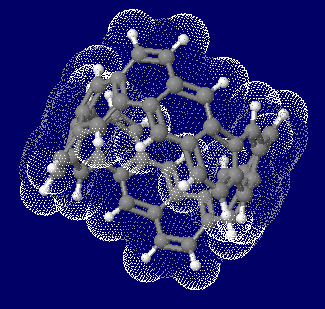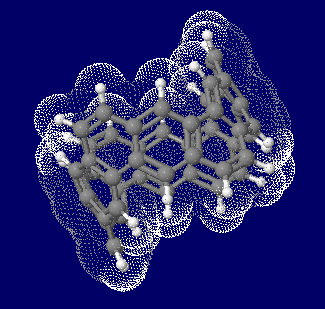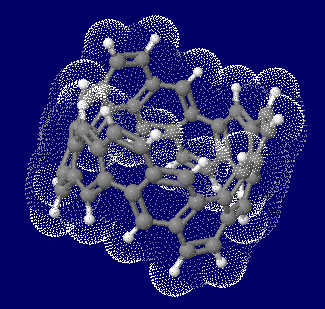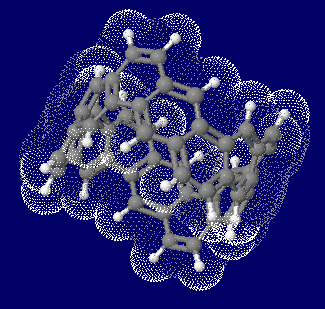
カーボンナノベルト(carbon nanobelt,CNB;還数12)の計算構造例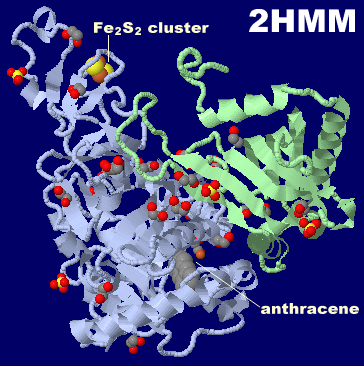
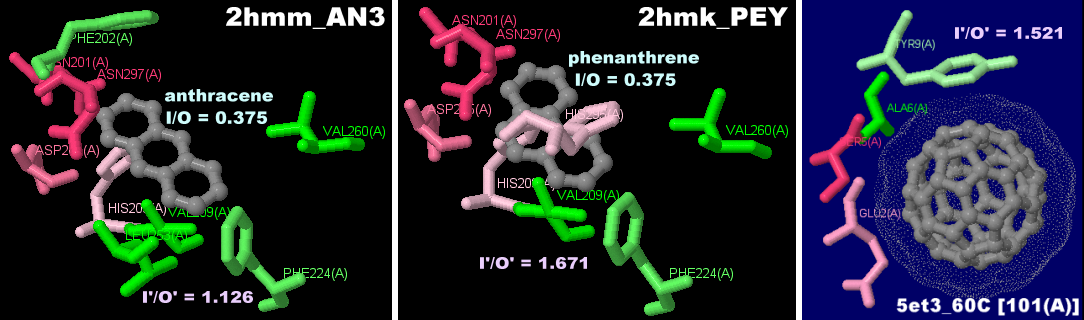
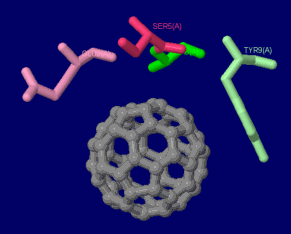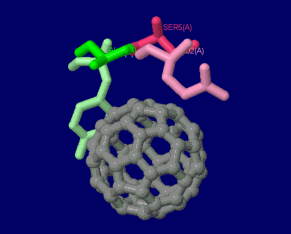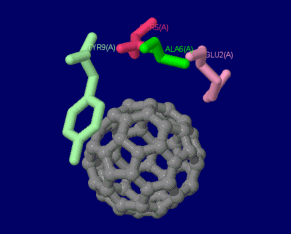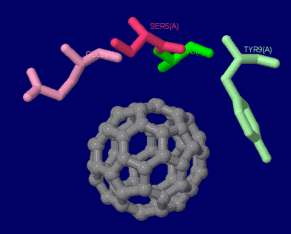
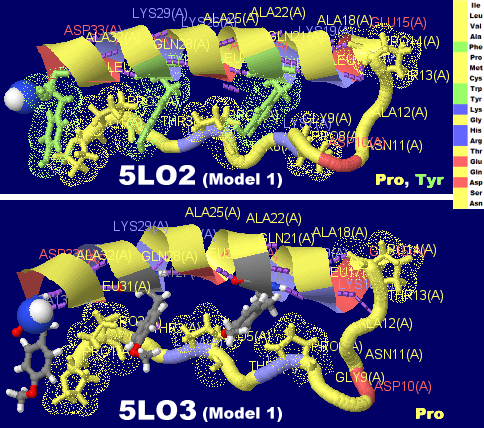
[左]アントラセンが結合したナフタレン-1,2-ジオキシゲナーゼ2hmm [中]参考PDBsumデータ [右]フラーレン結合デノボデザインペプチド5et3のPDBsumデータ
-
●アミノ色表示の凡例
ASP GLU CYS MET LYS ARG SER THR PHE TYR ASN GLN GLY LEU VAL ILE ALA TRP HIS PRO
●酸性・中性〈芳香族〉・塩基性アミノ酸区別表示の凡例
ASP GLU GLY ALA VAL LEU ILE CYS SER THR ASN GLN PRO MET PHE TYR TRP LYS ARG HIS
●極性〈酸性・塩基性〉・非極性(疎水性)アミノ酸区別
SER THR TYR CYS ASN GLN ASP GLU LYS ARG HIS GLY ALA VAL LEU ILE PHE PRO MET TRP
●疎水性インデックス順
ARG LYS ASN ASP GLN GLU HIS PRO TYR TRP SER THR GLY ALA MET CYS PHE LEU VAL ILE
●有機概念図I/O値順(特性基 R)
ASN SER ASP GLN GLU THR ARG HIS GLY LYS TYR TRP CYS MET PRO PHE ALA VAL LEU ILE
●等電点順
ASP GLU CYS ASN PHE GLN TYR SER MET TRP VAL GLY LEU ALA ILE THR PRO HIS LYS ARG
アミノ酸および特性基の親水性・疎水性 | Log Pをポケットに!


鉄硫黄クラスタータンパク質TtuA 5b4eとそのPDBsumデータ(他のFe4S4クラスターとの比較参照)
[Cytochrome P450 - Wikipedia,Ergocalciferol - Wikipedia]
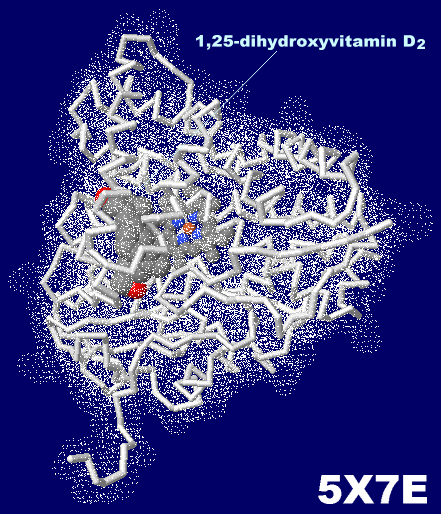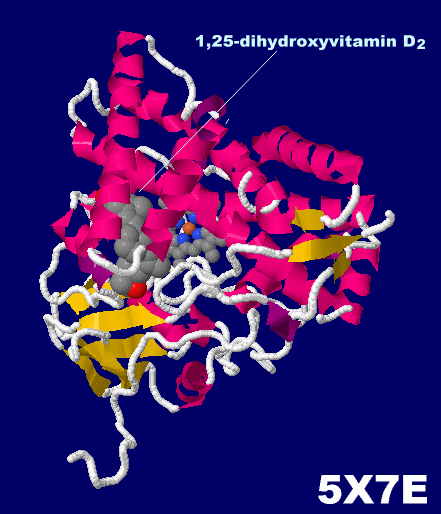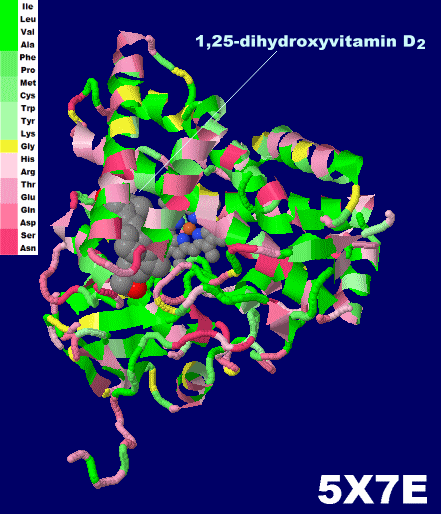
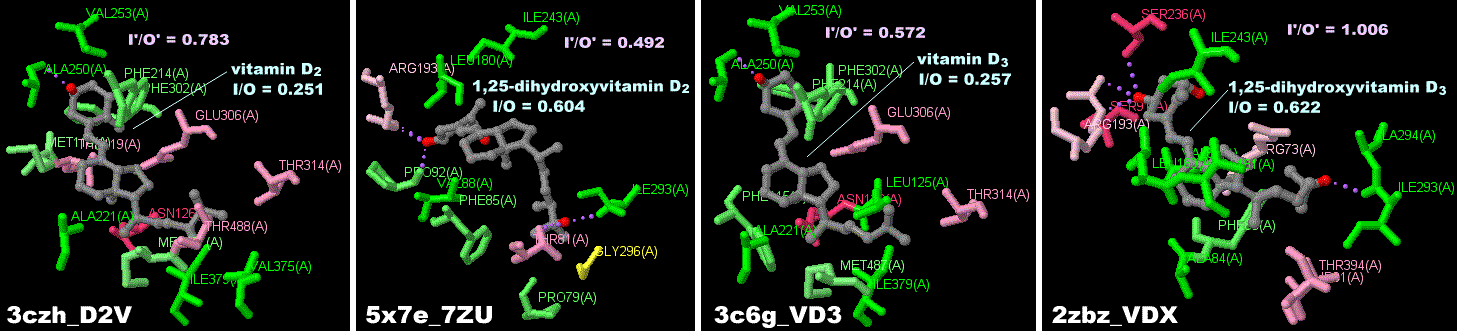
[左]1α,25-ジヒドロキシビタミンD2が結合したシトクロムP450 5x7e
[右]ビタミンD類が結合したP450のPDBsumデータ例
[Calcium pump - Wikipedia,Calcium ATPase - Wikipedia]


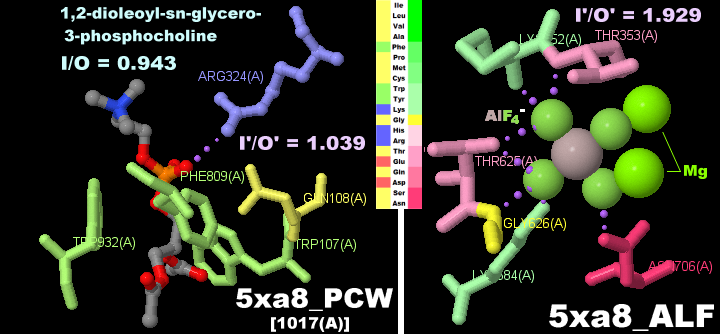
左からリン脂質,AlF4-,ADP,Ca2+が結合したカルシウムポンプ5xa8とそのリン脂質部分の例(Lys,Argのドット表面表示),およびPDBsumデータ(リン脂質とAlF4-)
※中央の図の右側はSPring-8プレスリリース(2017/05/04)参照
[Taste receptor - Wikipedia,TAS1R2 - Wikipedia]
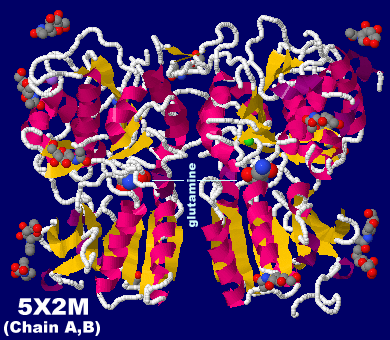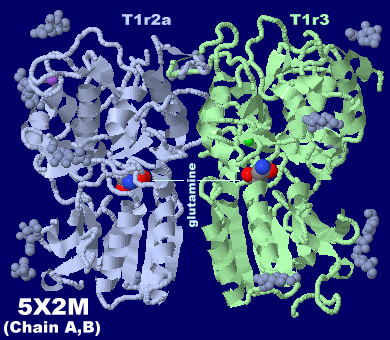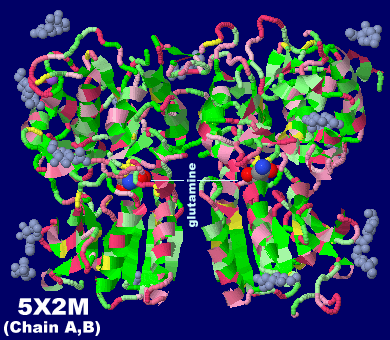
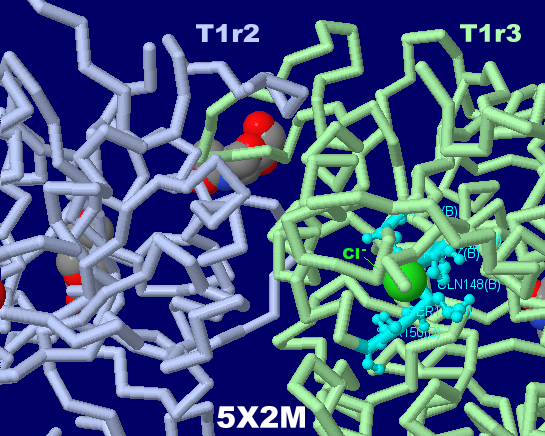

グルタミンと塩化物イオンが結合した味覚受容体T1r2-T1r3ヘテロ二量体5x2mのChain A・Bとその塩化物イオン周辺アミノ酸残基強調表示(eLife論文より),
およびそのPDBsumデータ(塩化物イオン;以下は他のリガンドについてのChain A・B・Dの比較)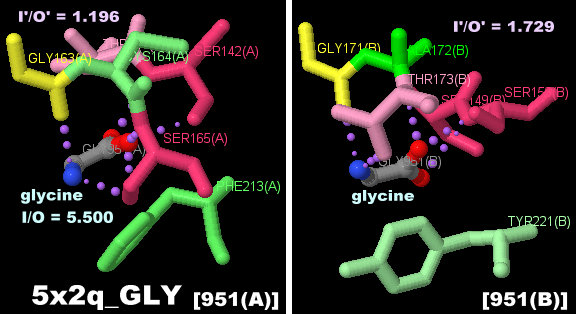


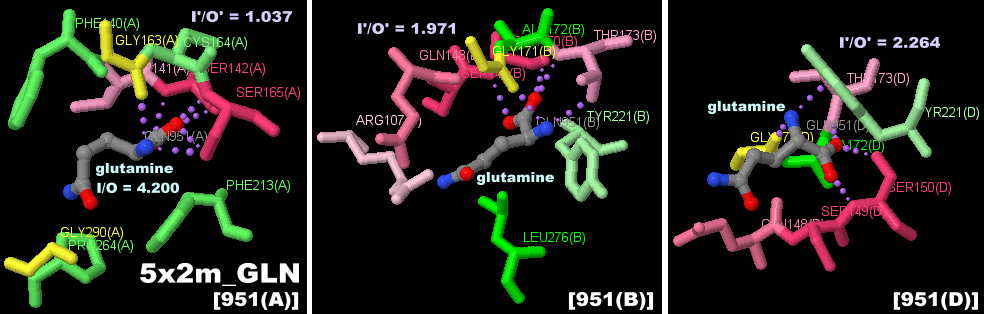
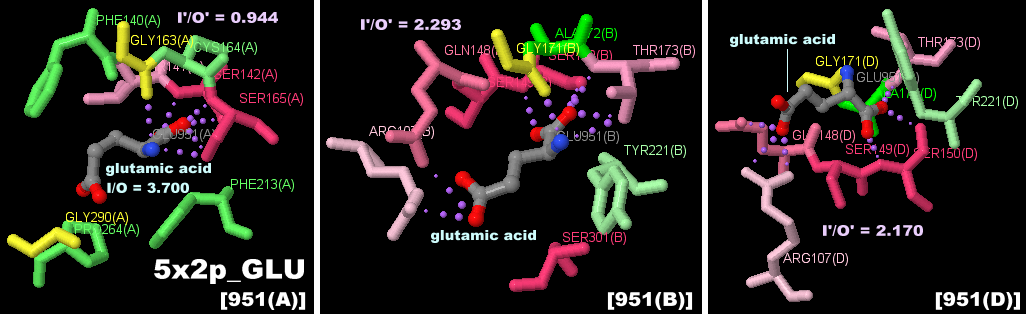
味覚受容体T1r2-T1r3ヘテロ二量体のPDBsumデータ(上から);5x2q(グリシン結合),5x2n(アラニン結合),5x2o(アルギニン結合),5x2m(グルタミン結合),5x2p(グルタミン酸結合)
メダカの味覚受容体T1r 5x2mのChain A・BのeLife論文およびJCCJ論文で言及されているアミノ酸残基の球棒・着色表示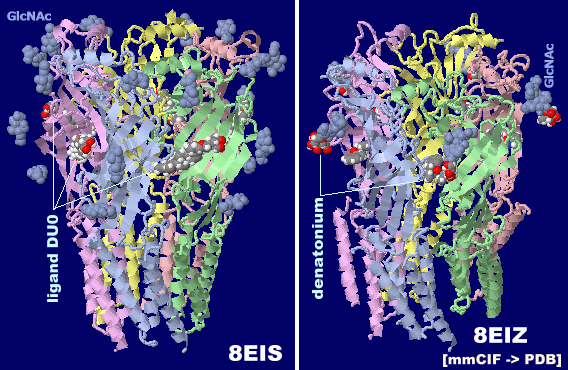

タコの走化性受容体CRT1 8eisとイカの化学触覚受容体CRT1 8eiz(cif→pdbデータ変換),およびそれぞれのPDBsumデータ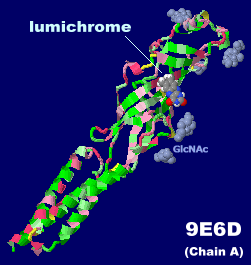
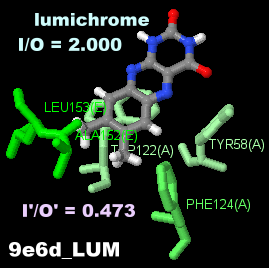
ルミクローム(ルミクロム,lumichrome)が結合したタコの化学触覚受容体CRT1 9e6dのChain AとPDBsumデータ
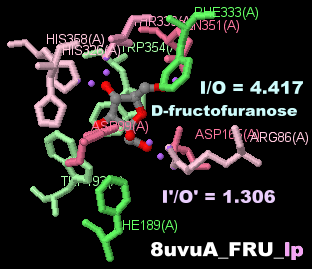
D-フルクトフラノース(重なりのD-フルクトピラノース削除)が結合したカイコの味覚受容体BmGr9 8uvuのChain AとそのPDBsumデータ


AGCGAリッチのDNA四重鎖構造例 5m2l
[Riboswitch - Wikipedia,Aptamer - Wikipedia]

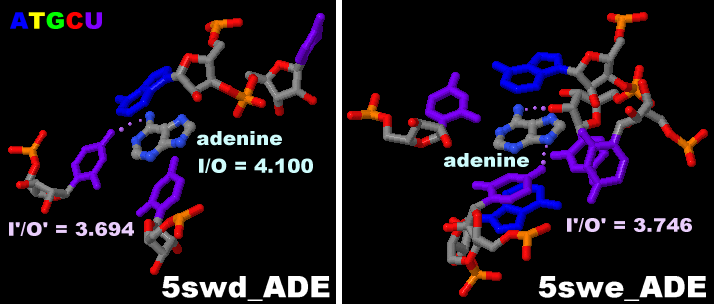
[左]アデニンが結合したアデニンリボスイッチアプタマードメイン5swe [右]5swd(アデニン結合途中)と5sweのPDBsumデータ
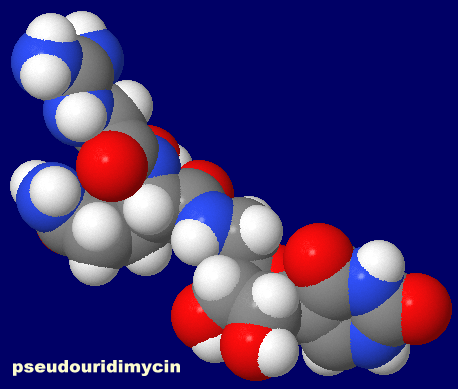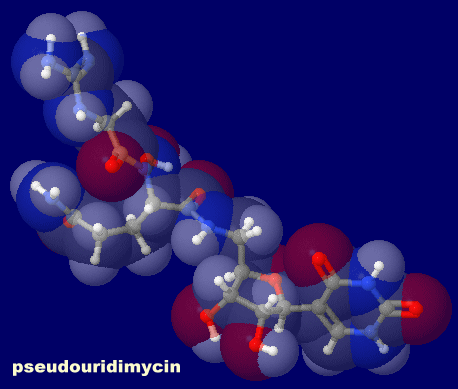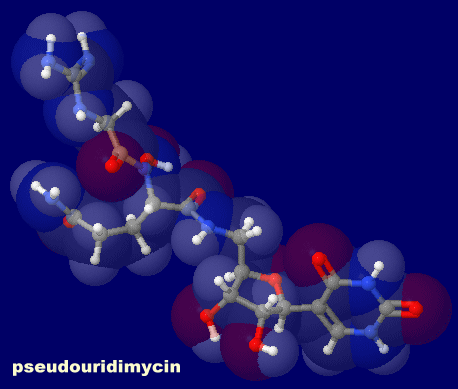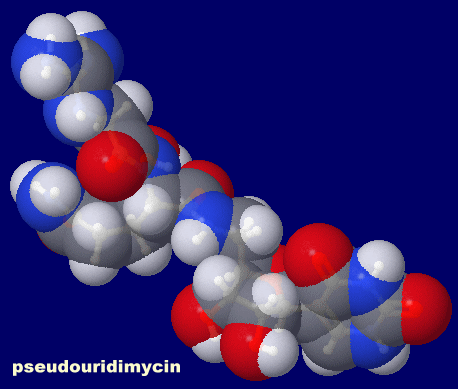


左からシュードウリジマイシン(pseudouridimycin,PUM)と同分子が結合したRNAポリメラーゼ5x21および同PDBsumデータ(アミノ酸残基・核酸塩基それぞれのI・O加算してI''/O''値算出)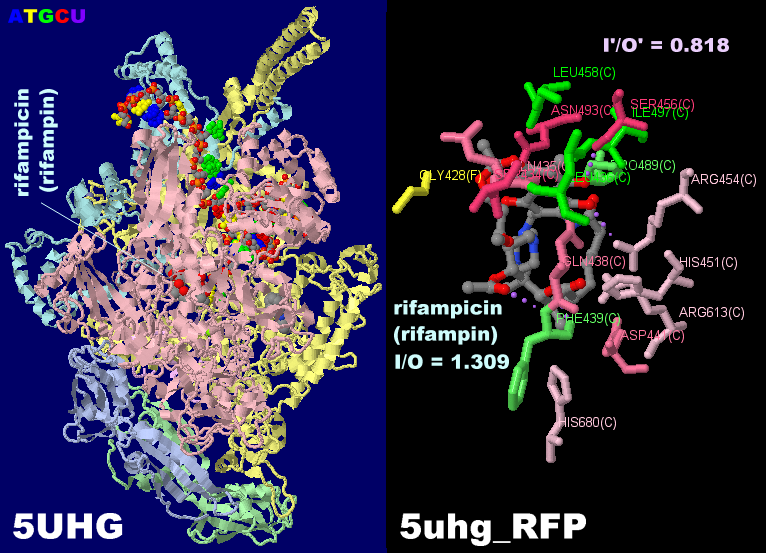
リファンピシン(リファンピン)が結合したRNAポリメラーゼ5uhgとそのPDBsumデータ
[Benzo(a)pyren - Wikipedia]

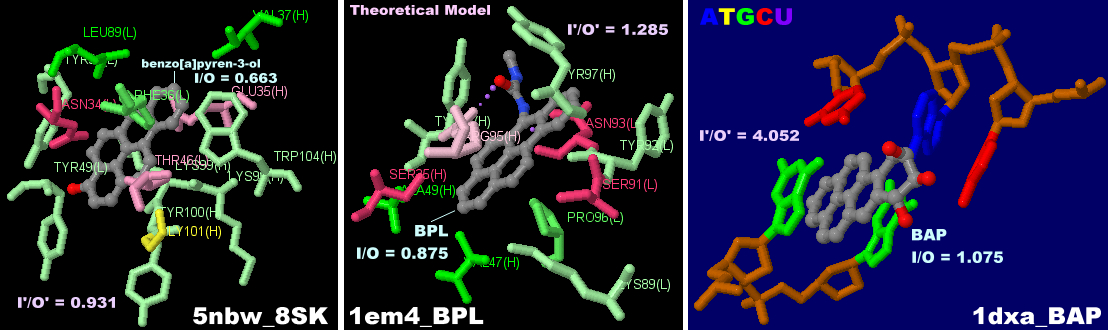
[左]3-ヒドロキシベンゾ[a]ピレンが結合したFabフラグメント22F12 5nbwのChain L・H
[右]5nbw,1em4,1dxaのPDBsumデータ比較