- 植物科学:植物フィトクロムBの非対称な二量体構造(Nature ハイライト,2022/04/07)
- Molecular ‘blueprint’ illuminates how plants perceive light(Washington University in St. Louis,2022/03/30)
- フィトクロム - Wikipedia | フィコシアノビリン
- フィトクロムB - 光合成事典 | フィコシアノビリン
[Phytochrome - Wikipedia,Phycocyanobilin - Wikipedia]


フィトクロモビリン(phytochromobilin;PΦB)が結合したシロイヌナズナのフィトクロムB 7rzwのChain AとそのPDBsumデータ
-
●アミノ色表示の凡例
ASP GLU CYS MET LYS ARG SER THR PHE TYR ASN GLN GLY LEU VAL ILE ALA TRP HIS PRO
●酸性・中性〈芳香族〉・塩基性アミノ酸区別表示の凡例
ASP GLU GLY ALA VAL LEU ILE CYS SER THR ASN GLN PRO MET PHE TYR TRP LYS ARG HIS
●極性〈酸性・塩基性〉・非極性(疎水性)アミノ酸区別
SER THR TYR CYS ASN GLN ASP GLU LYS ARG HIS GLY ALA VAL LEU ILE PHE PRO MET TRP
●疎水性インデックス順
ARG LYS ASN ASP GLN GLU HIS PRO TYR TRP SER THR GLY ALA MET CYS PHE LEU VAL ILE
●有機概念図I/O値順(特性基 R)
ASN SER ASP GLN GLU THR ARG HIS GLY LYS TYR TRP CYS MET PRO PHE ALA VAL LEU ILE
●等電点順
ASP GLU CYS ASN PHE GLN TYR SER MET TRP VAL GLY LEU ALA ILE THR PRO HIS LYS ARG
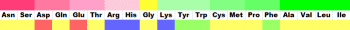
アミノ酸および特性基の親水性・疎水性 | Log Pをポケットに!
[AMPA receptor - Wikipedia]

黄色ブドウ球菌の薬物排出ポンプNorA 7lo8のChain Z(NorA)と結合しているFab36 CDRH3ループ(軽鎖Chain L中)
[Fidaxomicin - Wikipedia]


フィダキソマイシン(fidaxomicin)が結合したディフィシレ菌のRNAポリメラーゼ7l7bのChain DとそのPDBsumデータ
[Histone acetyltransferaser - Wikipedia,p300-CBP coactivator family - Wikipedia]


DS17701585が結合したヒストンアセチルトランスフェラーゼp300(EP300/CBPファミリー)7vi0のChain AとそのPDBsumデータ

DS-9300類縁体が結合したヒストンアセチルトランスフェラーゼp300(EP300/CBPファミリー)8gzcのChain AとそのPDBsumデータ
[Photolyase - Wikipedia]


DNA光回復酵素(DNAフォトリアーゼ)7vj1とそのPDBsumデータ(7vj0との比較),“Arg378, Asn403 and Asp409”は論文参照
[Ionotropic glutamate receptor - Wikipedia,Cyclothiazide - Wikipedia]



[左]血圧降下薬・利尿薬シクロチアジドが結合したイオンチャネル型グルタミン酸受容体(iGluR)7tnpのChain A
[中・右]別研究のiGluR(AMPA受容体;抗てんかん薬ペランパネル結合) 5l1fとそのPDBsumデータ

オピオイド受容体の主要なクラス例μ 4dkl,κ 4djh,δ 4ej4,および関連するノシセプチンオピオイド受容体(NOP) 4ea3 → GPCR - 2012年ノーベル化学賞

スボレキサント結合の6to7(OX1受容体)と6tpj(OX2受容体,どちらもChain A)とそれらのPDBsumデータ(後者はスボレキサントのH原子付加)

レンボレキサント結合のOX1受容体6totとそのPDBsumデータ
EMPA結合のOX2受容体5ws3とそのPDBsumデータ


[左]ナルフラフィン(nalfurafine) [中・右]ナルフラフィンが結合したκオピオイド受容体7yitのChain AとそのPDBsumデータ(JmolデータはGPCRに)

アゴニストMP1104とナノボディが結合したκオピオイド受容体6b73のChain A・DとそのPDBsumデータ
参考(オレキシン構造例):オレキシンA 1r02

超好熱性アーキアのtRNA 7vnvのChain A(空間充填表示がリン酸化ウリジンUp47,小さい球表示がその他のヘテロ原子)
[Hexazine - Wikipedia]

ヘキサジン(hexazine)
[cGMP-specific phosphodiesterase type 5 - Wikipedia]


ホスホジエステラーゼ5(PDE5)7faqとそのPDBsumデータ