- 掲載記事より
- 宮崎雅雄・上野山怜子・西川俊夫,「ネコのマタタビ行動」 → Jmolトピック
- 近藤健二,「新型コロナウィルス感染症による嗅覚障害」 → 新型コロナウイルス(SARS-CoV-2|COVID-19)情報
- GPCR - 2012年ノーベル化学賞(本サイト)
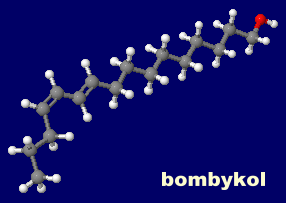
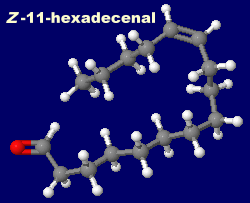
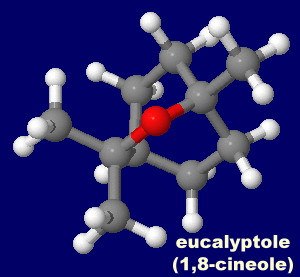
左から ボンビコール(bombykol),Z-11-hexadecenal(Z-11-ヘキサデセナール),ユーカリプトール(1,8-シネオール,1,8-cineole)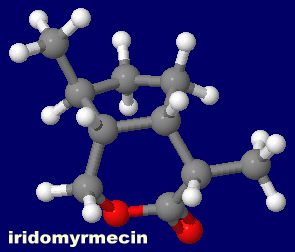
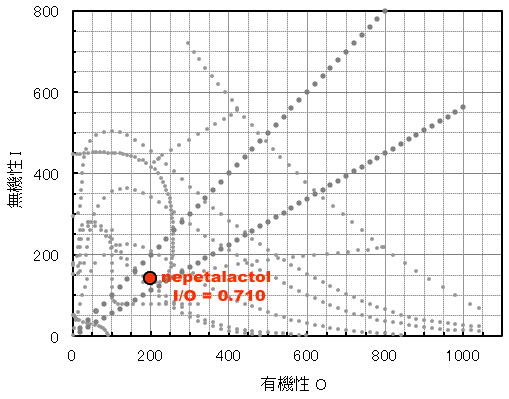
[左]イリドミルメシン(iridomyrmecin;マタタビラクトンの例) [中・右]cis-trans-ネペタラクトール(cis-trans-nepetalactol)とその有機概念図上の位置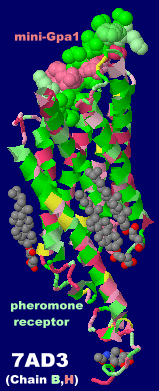
[左]フェロモン受容体構造例のChain B・H(フェロモン受容体〈クラスD GPCR,Ste2〉)
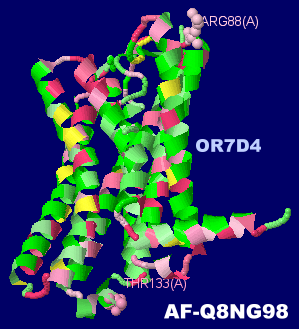
[右]OR7D4のAlphaFold2構造(UniProt id Q8NG98),ARG88・Thr133を球表示(特集p.416図1 参照)
-
●アミノ色表示の凡例
ASP GLU CYS MET LYS ARG SER THR PHE TYR ASN GLN GLY LEU VAL ILE ALA TRP HIS PRO
●酸性・中性〈芳香族〉・塩基性アミノ酸区別表示の凡例
ASP GLU GLY ALA VAL LEU ILE CYS SER THR ASN GLN PRO MET PHE TYR TRP LYS ARG HIS
●極性〈酸性・塩基性〉・非極性(疎水性)アミノ酸区別
SER THR TYR CYS ASN GLN ASP GLU LYS ARG HIS GLY ALA VAL LEU ILE PHE PRO MET TRP
●疎水性インデックス順
ARG LYS ASN ASP GLN GLU HIS PRO TYR TRP SER THR GLY ALA MET CYS PHE LEU VAL ILE
●有機概念図I/O値順(特性基 R)
ASN SER ASP GLN GLU THR ARG HIS GLY LYS TYR TRP CYS MET PRO PHE ALA VAL LEU ILE
●等電点順
ASP GLU CYS ASN PHE GLN TYR SER MET TRP VAL GLY LEU ALA ILE THR PRO HIS LYS ARG
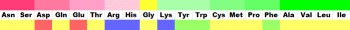
アミノ酸および特性基の親水性・疎水性 | Log Pをポケットに!
[Cellulase - Wikipedia]
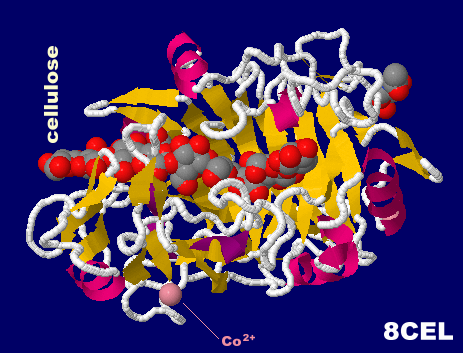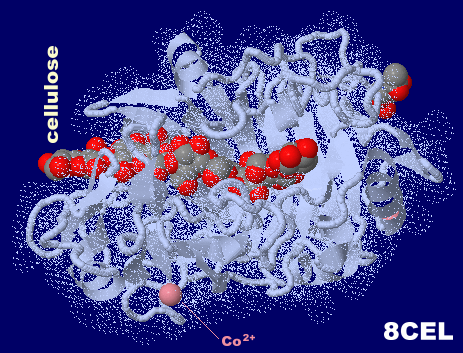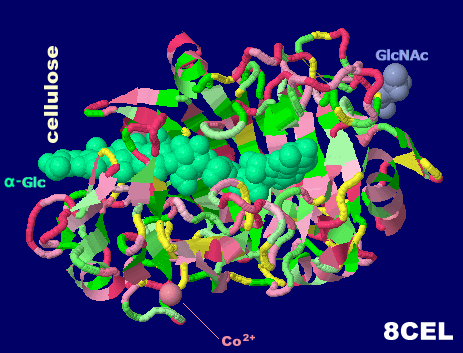
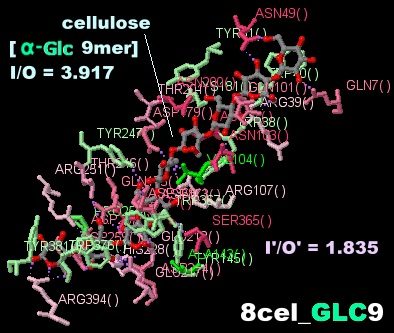
セルロースが結合したセルラーゼ 8cel(現在は削除されている理論モデルの構造;上掲記事では7cel掲載)とそのPDBsumデータ
[Epidermal growth factor receptor - Wikipedia]
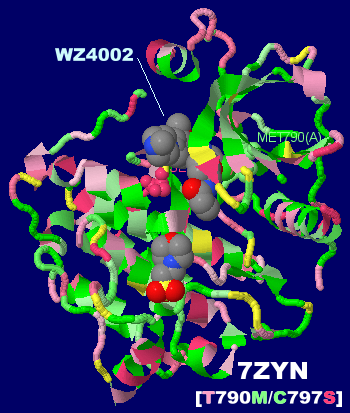
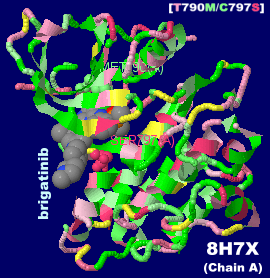
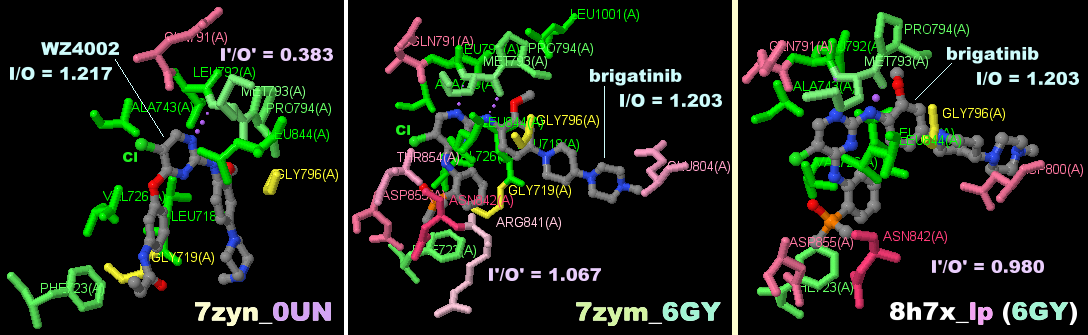
[左]阻害剤WZ4002が結合した上皮成長因子受容体(EGFR)7zyn [中]ブリグチニブ(brigatinib)が結合したEGFR 8h7xのChain A
[右]7zyn(WZ4002結合),7zym(ブリグチニブ結合),8h7x(同)のPDBsumデータ比較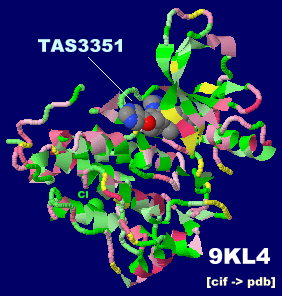
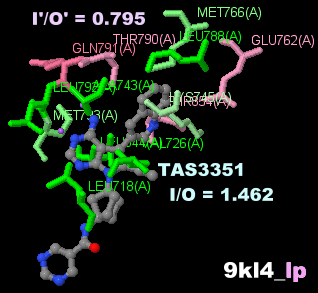
阻害剤TAS3351が結合したEGFR 9kl4とPDBsumデータ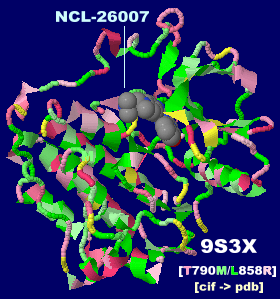
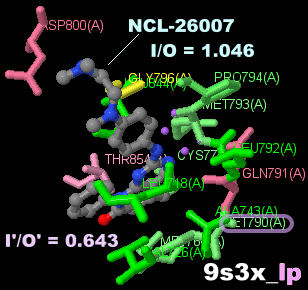
阻害剤NCL-26007が結合したEGFR 9s3xとPDBsumデータ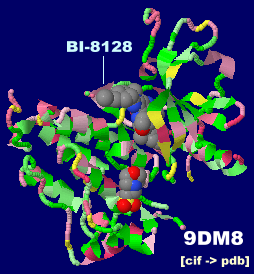
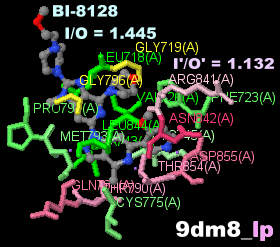
阻害剤BI-8128が結合したEGFR 9dm8とPDBsumデータ

 ★「北海道石」ほかを日本コンピュータ化学会@サイエンスアゴラ2023(テレコムセンタービル,2023/11/18-19)でブース展示し,11/19の13:00-15:00には発見メンバーの田中陵二さんに解説していただきました。また,2024年1月の同石も紹介する「石は元素の案内人」(福音館書店)に続き,2024年8月にたくさんのふしぎ 2024年8月号「光る石 北海道石 新鉱物Hokkaidoiteはっけん記」(同)が発売されました。
★「北海道石」ほかを日本コンピュータ化学会@サイエンスアゴラ2023(テレコムセンタービル,2023/11/18-19)でブース展示し,11/19の13:00-15:00には発見メンバーの田中陵二さんに解説していただきました。また,2024年1月の同石も紹介する「石は元素の案内人」(福音館書店)に続き,2024年8月にたくさんのふしぎ 2024年8月号「光る石 北海道石 新鉱物Hokkaidoiteはっけん記」(同)が発売されました。
★田中陵二さんをゲストにお迎えして第118回サイエンスカフェにいがた『ふしぎな新鉱物「北海道石」を知ろう! 楽しもう!』(2024/04/22)を開催し,「北海道石」実物をご覧いただきました。
★「北海道石」を日本コンピュータ化学会@サイエンスアゴラ2024(テレコムセンタービル,2024/10/26-27)でブース展示しました。
[Benzo(ghi)perylene - Wikipedia]
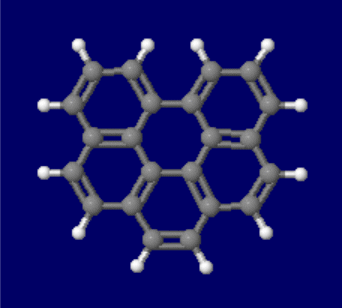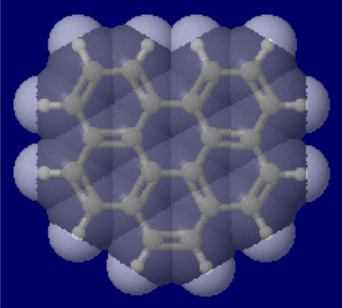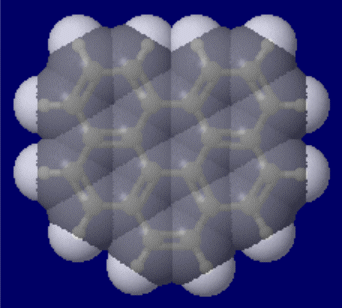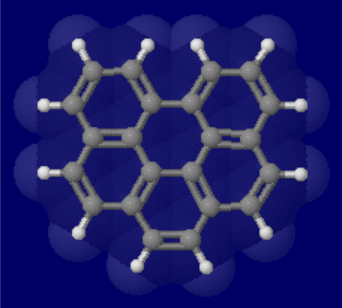

[左]ベンゾ[ghi]ペリレン(benzo[ghi]perylene) [右]同分子とコロネン(coronene)の同時表示(●は炭素)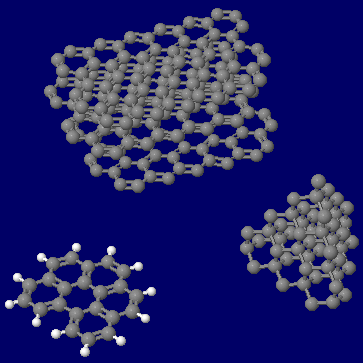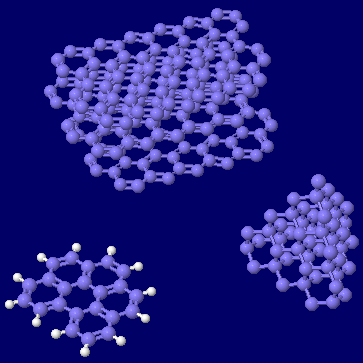

ベンゾ[ghi]ペリレンとダイヤモンドとグラファイトの同時表示(アニメgifで●は炭素)およびその3Dプリンタ模型(スタジオミダス製作)


左から 日本コンピュータ化学会@サイエンスアゴラ2023での北海道石展示,同解説パネル,ベンゾ[ghi]ペリレン・コロネン・ピセンを含む展示鉱石
※日本コンピュータ化学会2023秋季年会一般公開イベント(2023/11/26,高松市 レクザムホール)でも展示させていただきました。 → 会場写真
[Taurine - Wikipedia]

[左]タウリン(taurine,アミノエタンスルホン酸)
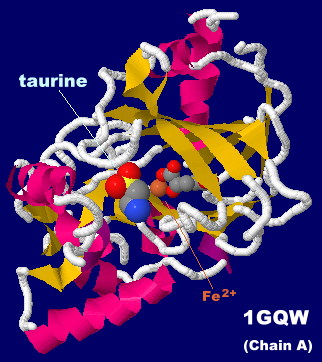
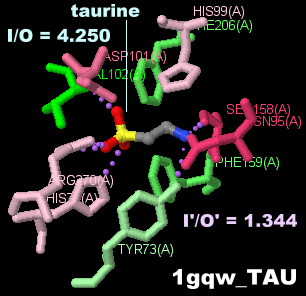
[中・右]タウリンが結合したタウリンジオキシゲナーゼ1gqwのChain AとPDBsumデータ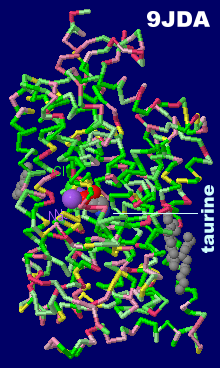
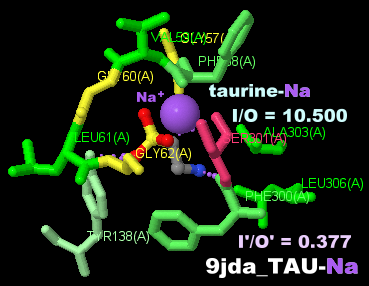
タウリントランスポーター9jdaとPDBsumデータ
[Kinase - Wikipedia]
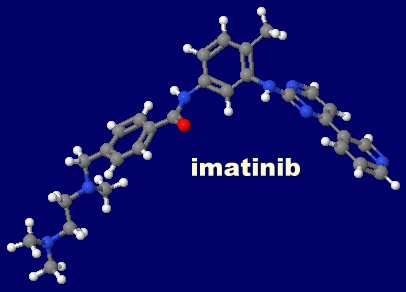
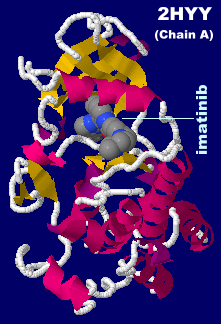
[左]イマチニブ(imatinib) [中・右]イマチニブ(imatinib)が結合したc-Ablタンパク質キナーゼ2hyyのChain AとPDBsumデータ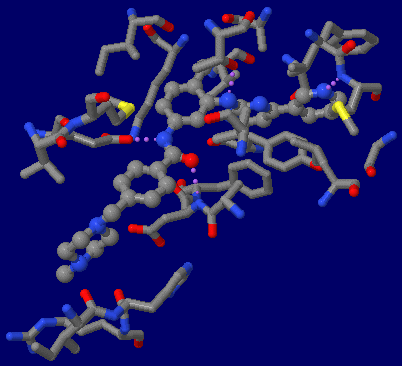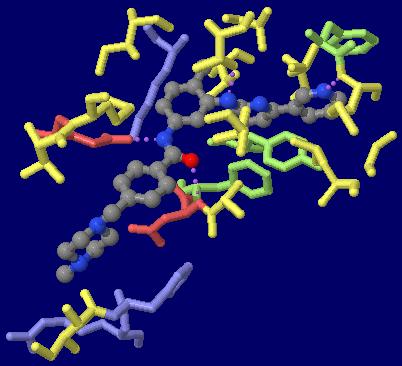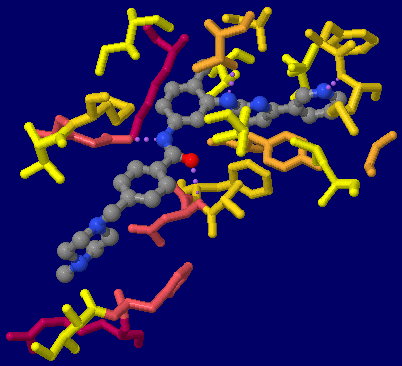

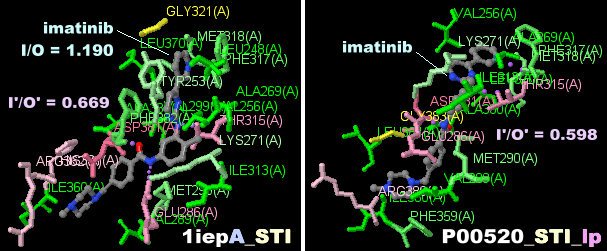
[左]分子標的薬の例イマチニブを含む1iepのPDBsumデータより
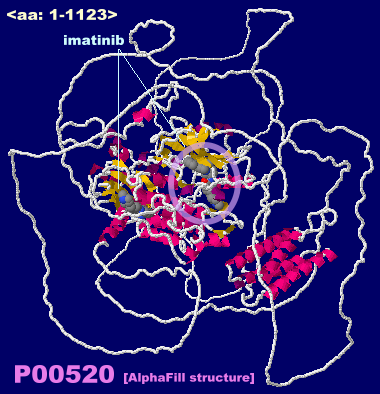
[中・右]イマチニブが結合したキノンレダクターゼc-Abl 1iepおよびAlphaFill構造 P00520(データ掲載ページ)のPDBsumデータと後者の全体構造
※「今月の分子 283:c-Ablタンパク質キナーゼとイマチニブ」ではAlphaFill構造 P00519紹介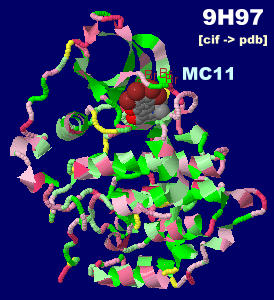
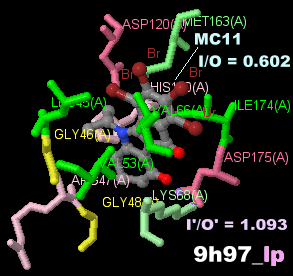
阻害剤MC11が結合したタンパク質キナーゼCK2 9h97とPDBsumデータ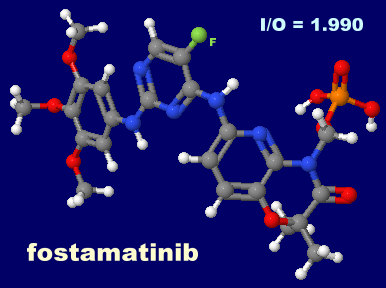
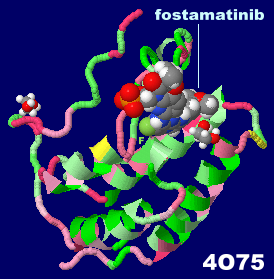
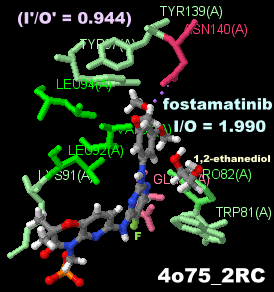
[左]ホスタマチニブ(fostamatinib) [中・右]参考データ:ホスタマチニブが結合したBRD4 4o75とPDBsumデータ

NF6-02構造7bqbのModel 1とNF4-04構造7bqcのModel 1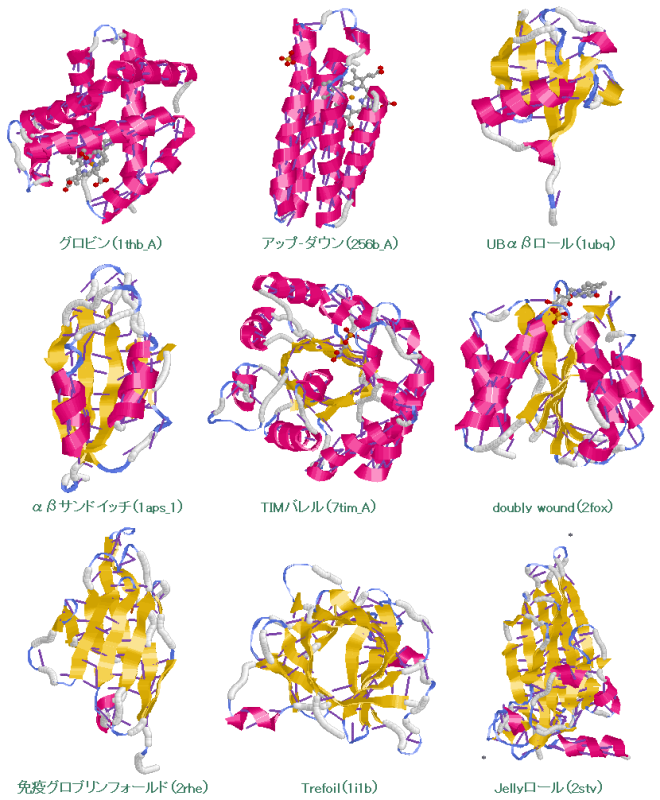
参考図:タンパク質の高次構造のいろいろ;スーパーフォールドの例(水素結合表示,リガンドは球棒モデル)
グロビン(1thbのChain A),アップ-ダウン(256bのChain A),UBαβロール(1ubq → ユビキチン #;2004年ノーベル化学賞)
αβサンドイッチ(1apsのModel 1),TIMバレル(7timのChain A),doubly wound(2fox)
免疫グロブリンフォールド(2rhe),Trefoil(1i1b),Jellyロール(2stv)
◎美宅成樹・榊佳之 編,「バイオインフォマティクス」,p.175,東京化学同人(2003)
◎後藤祐児・高橋聡,『タンパク質フォールディングの昼と夜』,現代化学,2004年9月号,pp.26-33,東京化学同人
→ 生体分子のかたちの不思議
[Tiratricol - Wikipedia]
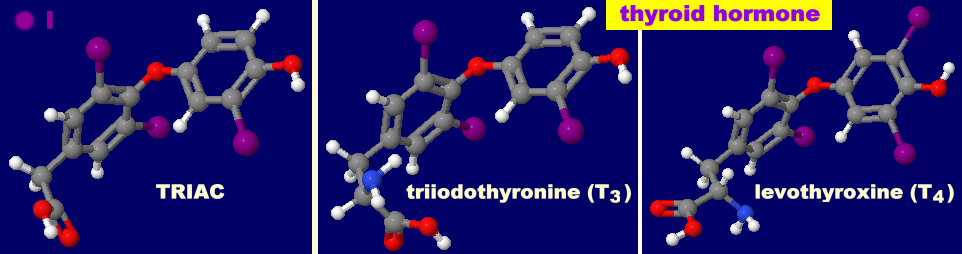
[左]TRIAC(tiratricol)および甲状腺ホルモンのトリヨードサイロニン(triiodothyronine,T3)とレボチロキシン(levothyroxine,T4)
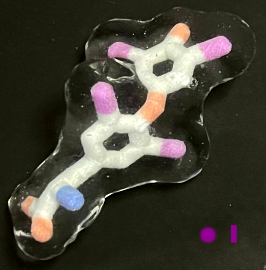
[右]レボチロキシン(levothyroxine,T4)の川上モデル(H原子は略)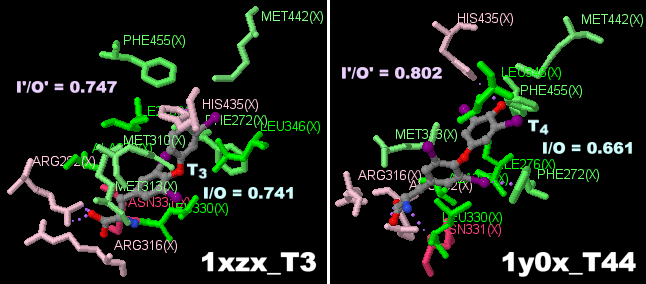
トリヨードサイロニン(T3)結合タンパク質1xzxおよびレボチロキシン(T4)結合タンパク質1y0xのPDBsumデータ
[Arsenite oxidase - Wikipedia]
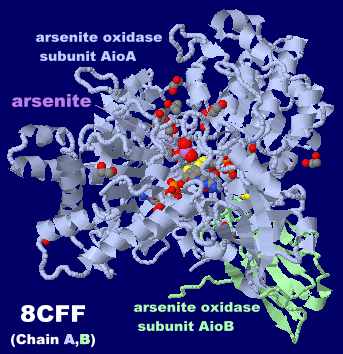
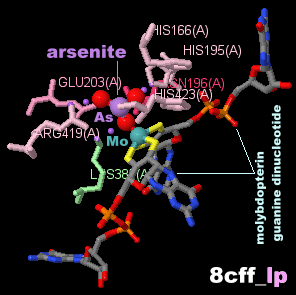
亜ヒ酸が結合した亜ヒ酸オキシダーゼ8cffのChain A・BとそのPDBsumデータ


[左]抗CRISPRファミリーAcrIF7 のModel 1 [右]天然タンパク質におけるアミノ酸使用のルール(東京大学プレスリリース図3を参考にした着色例)