- Molecular insights into the endoperoxide formation by Fe(II)/��-KG-dependent oxygenase NvfI�iNature Communications�C2012/07/20�j
- �w���y�f�֘A�ʌ����F�����w�F�A���J���C�h�����y�f�̍\���ƃX�[�p�[�I�L�V�h�@�\ �iNature �n�C���C�g�C2025/04/17�j [NEW!]
- Chanoclavine synthase operates by an NADPH-independent superoxide mechanism�iNature�C2025/03/05�j [NEW!]�@��2025/01/01���J9jdb�E9jdc�i���f�j
- �Q�l�F�A���J���C�h - ���f���猩�鐶���̐��E�i�{�T�C�g�j
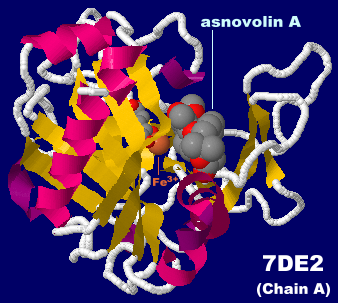
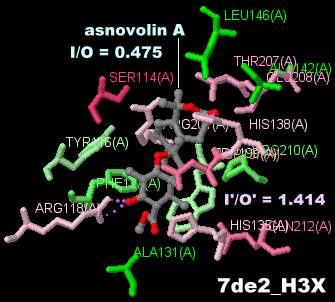
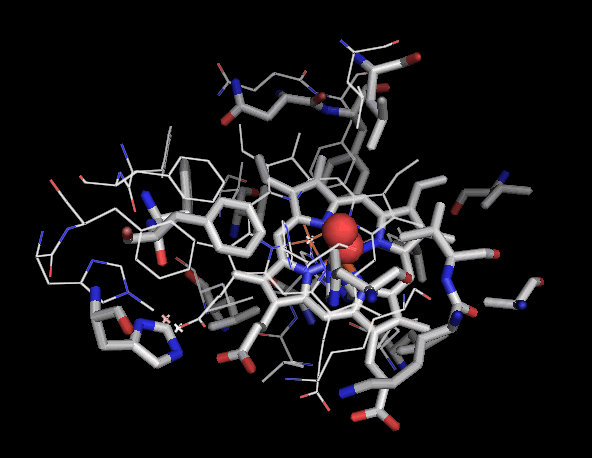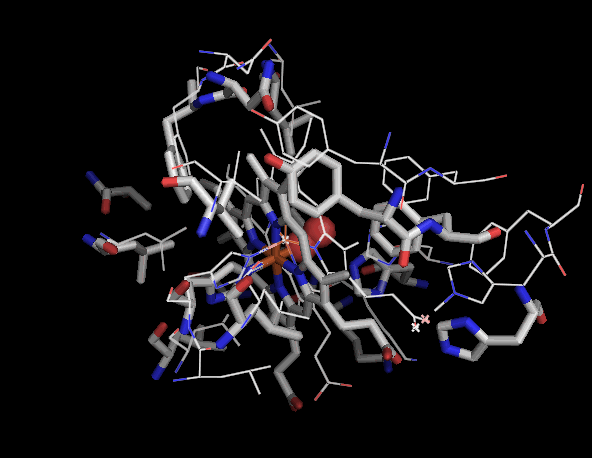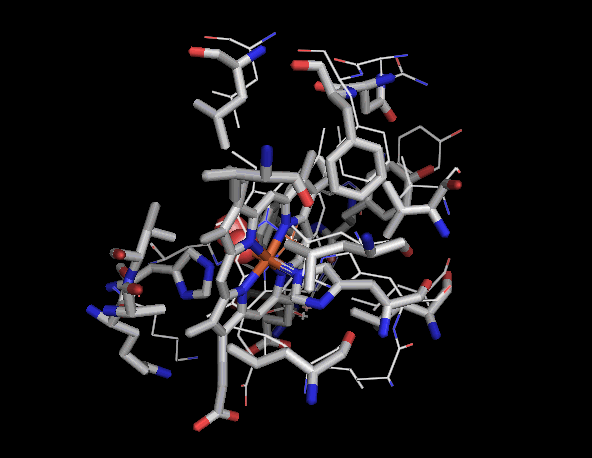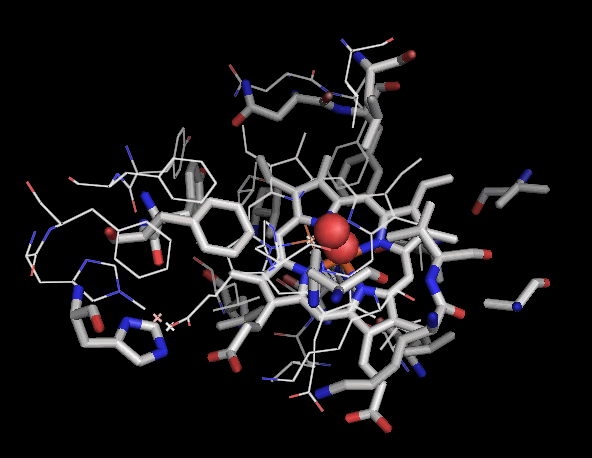
asnovolin A������������w���S�_�f�Y���y�fNvfI 7de2��Chain A��PDBsum�f�[�^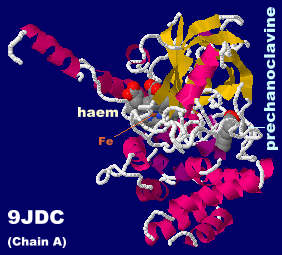
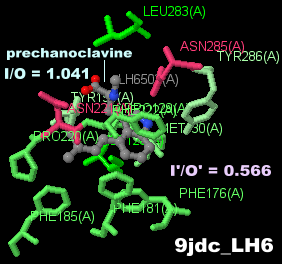
prechanoclavine�iPCC�j�����������A���J���C�h�����Ɋ֗^����w���y�f�i�J�^���[�[EasC�j 9jdc��Chain A��PDBsum�f�[�^
-
���A�~�m�F�\���̖}��
ASP�@GLU�@CYS�@MET�@LYS�@ARG�@SER�@THR�@PHE�@TYR�@ASN�@GLN�@GLY�@LEU�@VAL�@ILE�@ALA�@TRP�@HIS�@PRO
���_���E�����q�F�����r�E����A�~�m�_��ʕ\���̖}��
ASP�@GLU�@GLY�@ALA�@VAL�@LEU�@ILE�@CYS�@SER�@THR�@ASN�@GLN�@PRO�@MET�@PHE�@TYR�@TRP�@LYS�@ARG�@HIS
���ɐ��q�_���E����r�E��ɐ��i�a�����j�A�~�m�_���
SER�@THR�@TYR�@CYS�@ASN�@GLN�@ASP�@GLU�@LYS�@ARG�@HIS�@GLY�@ALA�@VAL�@LEU�@ILE�@PHE�@PRO�@MET�@TRP
���a�����C���f�b�N�X��
ARG�@LYS�@ASN�@ASP�@GLN�@GLU�@HIS�@PRO�@TYR�@TRP�@SER�@THR�@GLY�@ALA�@MET�@CYS�@PHE�@LEU�@VAL�@ILE
���L�@�T�O�}I/O�l���i������ R�j
ASN�@SER�@ASP�@GLN�@GLU�@THR�@ARG�@HIS�@GLY�@LYS�@TYR�@TRP�@CYS�@MET�@PRO�@PHE�@ALA�@VAL�@LEU�@ILE
�����d�_��
ASP�@GLU�@CYS�@ASN�@PHE�@GLN�@TYR�@SER�@MET�@TRP�@VAL�@GLY�@LEU�@ALA�@ILE�@THR�@PRO�@HIS�@LYS�@ARG
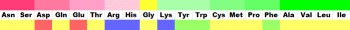
�A�~�m�_����ѓ�����̐e�����E�a���� �b Log P���|�P�b�g�ɁI
�����\���̓��^���n�C�h���[�g�imethanehydrate�j
�i�{�L�����ʃy�[�W�Ɉڂ��C����̍X�V�͂�����ōs���܂��j
�mAMPA receptor - Wikipedia�n
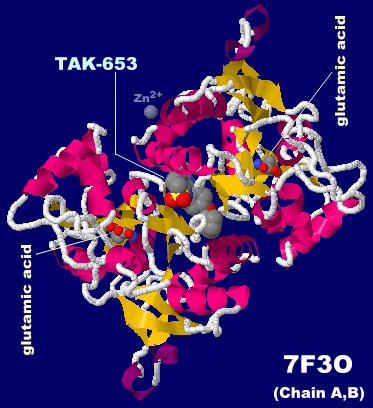
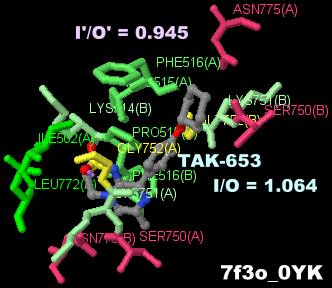
TAK-653����������AMPA���Z�v�^�[ 7f3o��Chain A�EB�Ƃ���PDBsum�f�[�^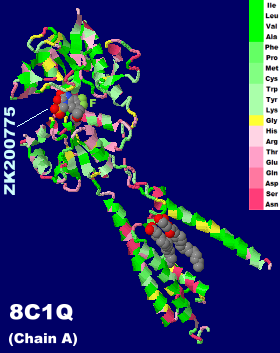
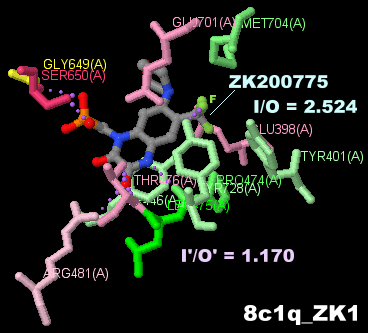
�A���^�S�j�X�gZK200775�����������J���V�E�����ߐ�AMPA�^�O���^�~���_��e�� 8c1q��Chain A�Ƃ���PDBsum�f�[�^

�����W�f�[�^�͍��������Y������Ă��������܂����B
�mRibonuclease P - Wikipedia�CRNase MRP - Wikipedia�n
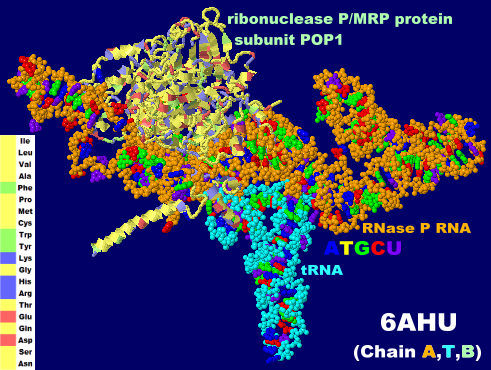
���{�k�N���A�[�[P��tRNA�̕����� 6ahu��Chain A�i���{�k�N���A�[�[P�j�ET�itRNA�j�EB�i�^���p�N���̃T�u���j�b�gPop1�̂݁j
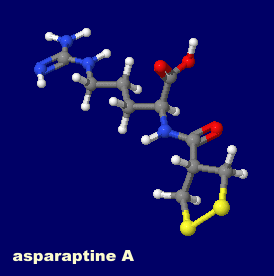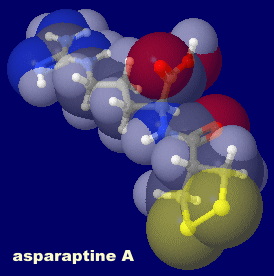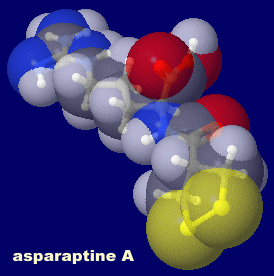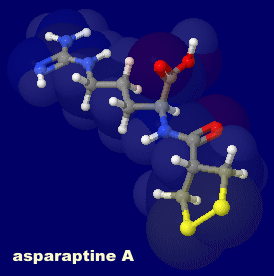
�A�X�p���v�`��A�iasparaptine A�GI/O = 2.107�j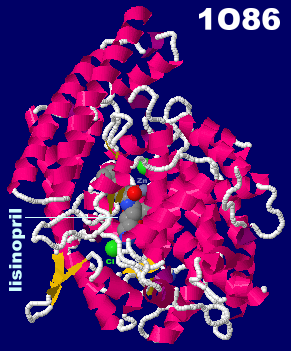
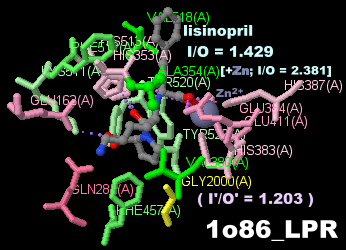
�Q�l�F�j�Q�܃��V�m�v�����ilisinopril�j�����������q�g�A���W�I�e���V���ϊ��y�f 1o86�Ƃ���PDBsum�f�[�^
�mBatrachotoxin - Wikipedia�n
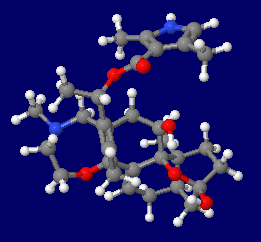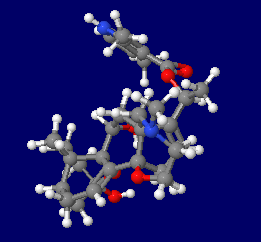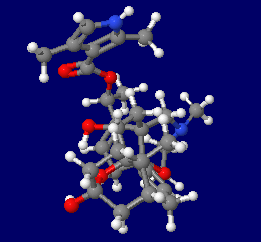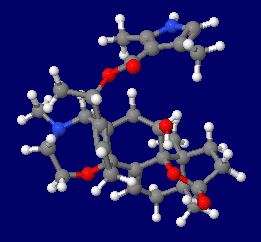
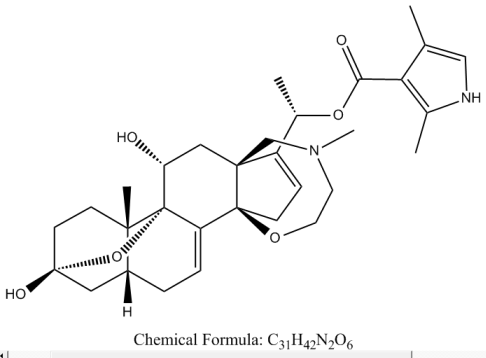
�o�g���R�g�L�V���ibatrachotoxin�CBTX�j�̕��q�\���ƍ\����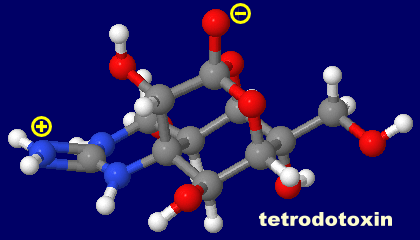
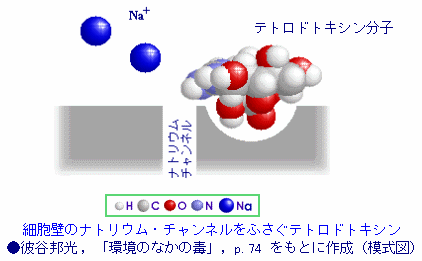
�Q�l�}�F �t�O�Ńe�g���h�g�L�V���itetrodotoxin�CTTX�j�Ƃ��̍�p�@���͎��}
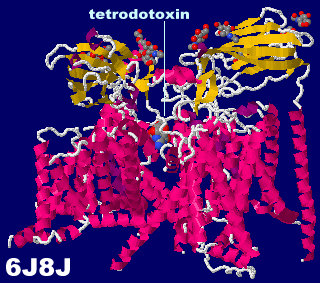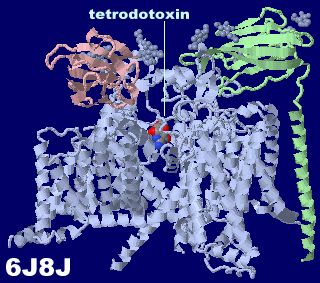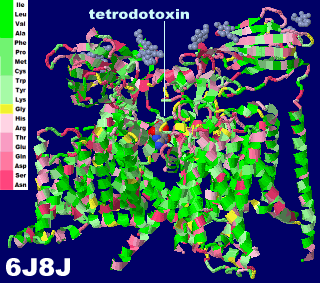
�e�g���h�g�L�V�������������d�ʈˑ����i�g���E���`���l��6j8j�Ƃ���PDBsum�f�[�^�@�� Jmol�g�s�b�N
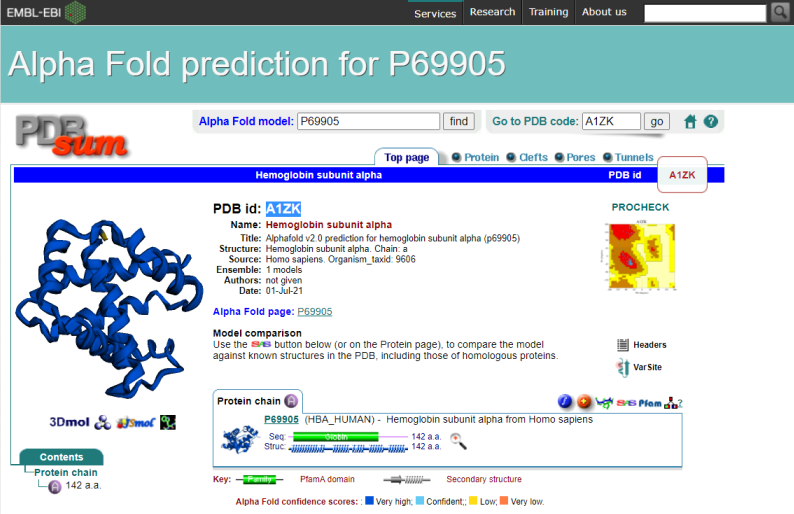
�m���nPDBsum�T�C�g�ŕ\�������P69905�\��
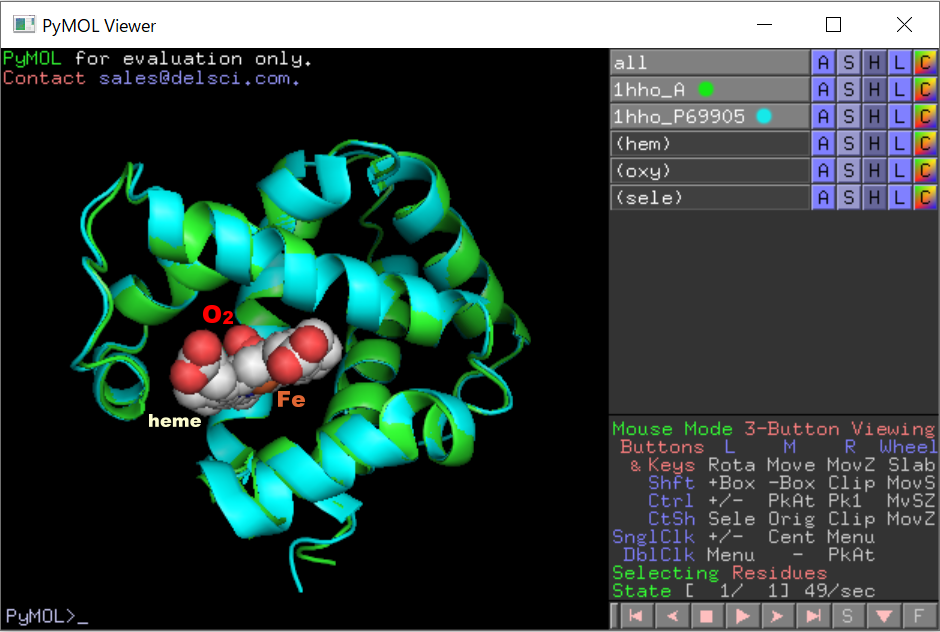
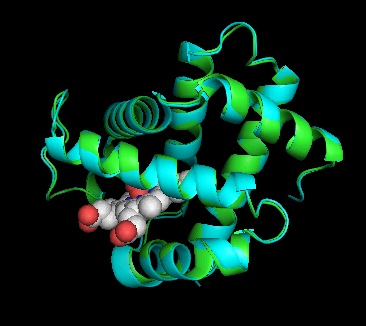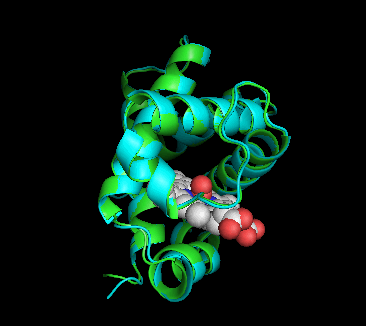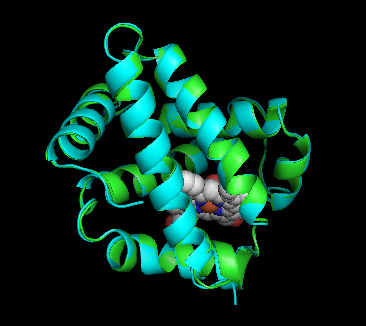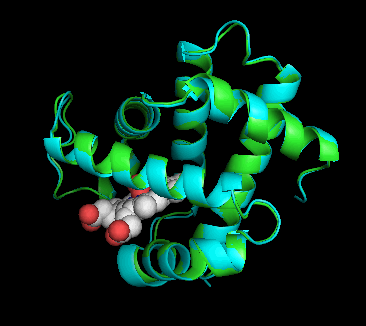
�m���E�E�nP69905�\���ƃq�g�I�L�V�w���O���r���̃w���O���r�����\����i1hho��Chain A�j��PyMOL�ɂ��d�ˍ��킹�摜�Ƃ��̉�]����
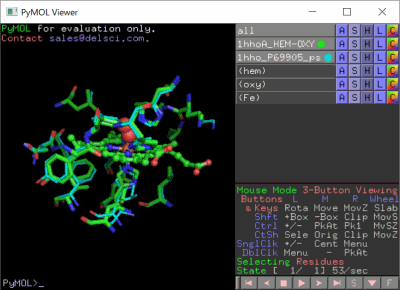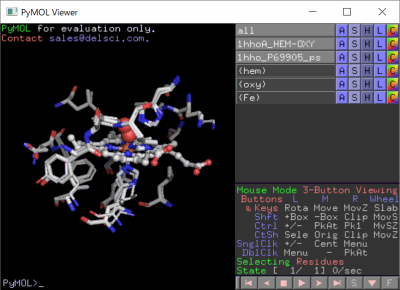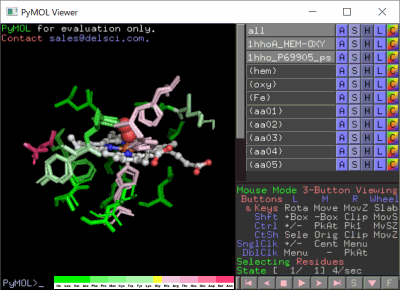
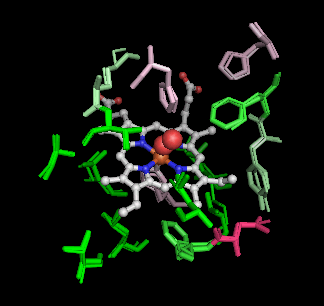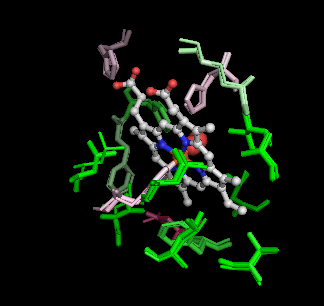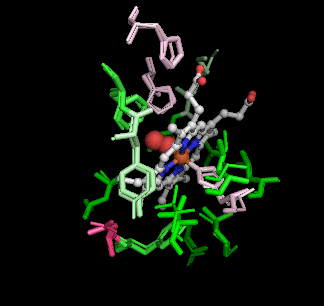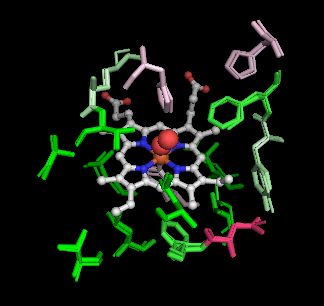
�m���E���n1hho��Chain A��PDBsum�f�[�^�i�����X�e�B�b�N�j��P69905�\�����炻��Ɠ����A�~�m�_�z����o�������́i�ׂ��X�e�B�b�N�j��PyMOL�ɂ��d�ˍ��킹�摜�Ƃ��̉�]����iI/O�l�����F�j
�m�E�n�Q�l�}�F�I�L�V�w���O���r��1hho�i�X�e�B�b�N�\���j�ƃf�I�L�V�w���O���r��2hhb�i�j���\���j��PyMOL�ɂ��d�ˍ��킹�\���i�����Chain A��PDBsum�f�[�^�j
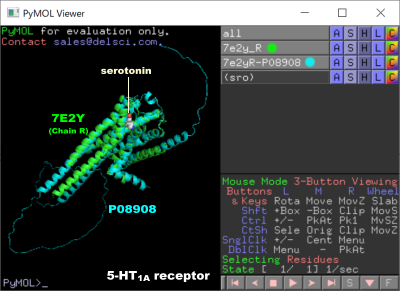
5-HT1A��e�̍\��P08908��7e2y�i�Z���g�j�������j��Chain R�̏d�ˍ��킹
�mPseudouridine - Wikipedia�n
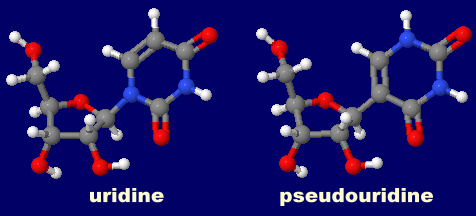
�m���n�E���W���iuridine�j�ƃV���[�h�E���W���ipseudouridine�j�̓����\��
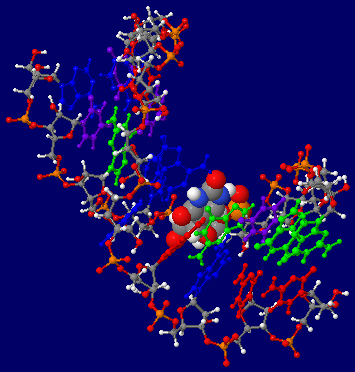
�m�E�n�Q�l�}�F ����ɃV���[�h�E���W���܂�RNA�̍\����i���q�ԍ�156�����GmRNA���N�`���Ƃ͖��W�j1lpw��Model 1
�mProtein tyrosine phosphatase - Wikipedia�CPTPN11 - Wikipedia�n
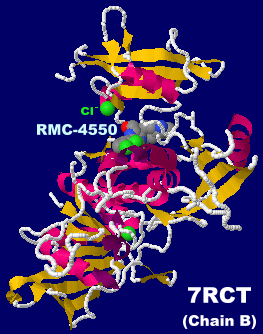
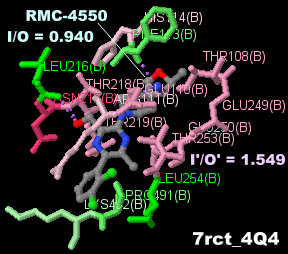
�j�Q��RMC-4550�����������^���p�N���`���V���z�X�t�@�^�[�[SHP2�iPTPN11�j 7rct��Chain B�Ƃ���PDBsum�f�[�^
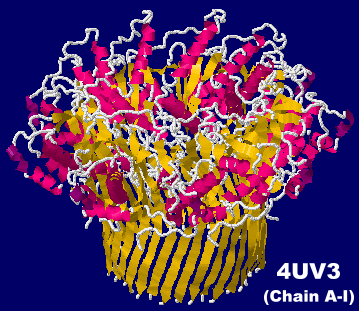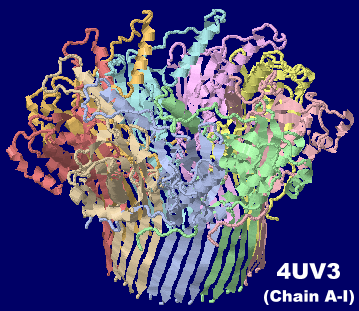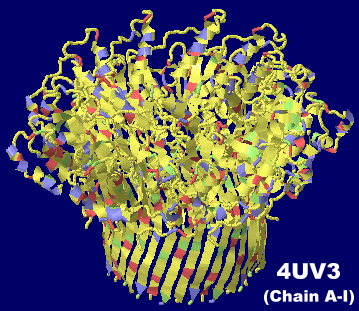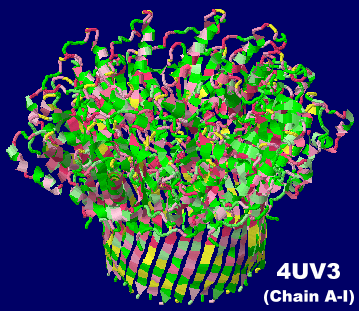
�咰��BL21(DE3)�̊O���^���p�N��CsgG 4uv3��Chain A-I