- ステビア含有天然甘味成分を合成する酵素の改良に成功 -希少甘味成分の蓄積向上に期待-(富山大学,2025/01/10) [PDF]
- 引用7es1の論文:Catalytic flexibility of rice glycosyltransferase OsUGT91C1 for the production of palatable steviol glycosides(Nature Communications,2021/12/02)
- グリコシルトランスフェラーゼ - Wikipedia
- 甘味物質の秘密(本サイト)
[Glycosyltransferase - Wikipedia]
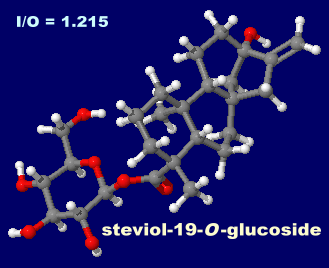
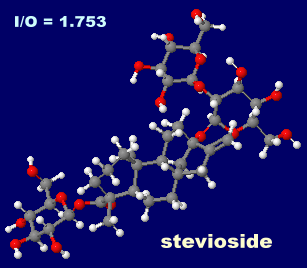
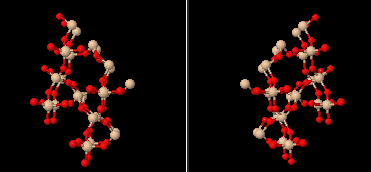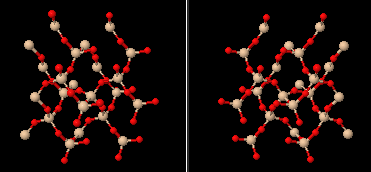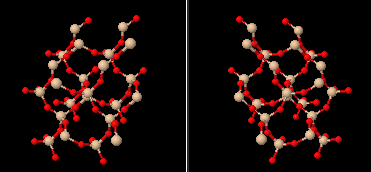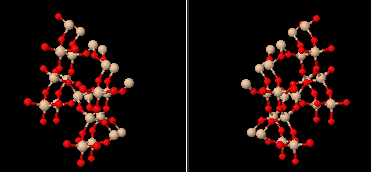
ステビオール-19-O-グルコシド(steviol-19-O-glucoside)とステビオサイド(stevioside;ステビア甘味成分の例)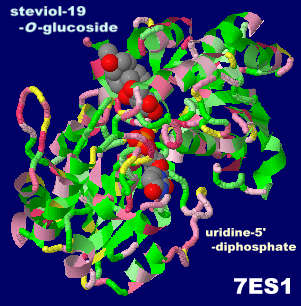
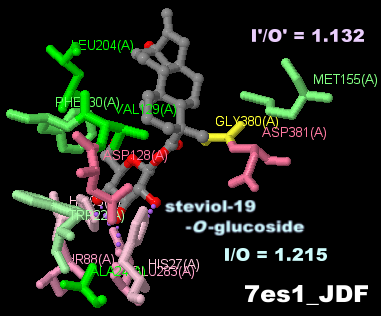
ステビオール-19-O-グルコシドが結合したイネ由来の糖転移酵素 OsUGT91C1 7es1とPDBsumデータ
-
●アミノ色表示の凡例
ASP GLU CYS MET LYS ARG SER THR PHE TYR ASN GLN GLY LEU VAL ILE ALA TRP HIS PRO
●酸性・中性〈芳香族〉・塩基性アミノ酸区別表示の凡例
ASP GLU GLY ALA VAL LEU ILE CYS SER THR ASN GLN PRO MET PHE TYR TRP LYS ARG HIS
●極性〈酸性・塩基性〉・非極性(疎水性)アミノ酸区別
SER THR TYR CYS ASN GLN ASP GLU LYS ARG HIS GLY ALA VAL LEU ILE PHE PRO MET TRP
●疎水性インデックス順
ARG LYS ASN ASP GLN GLU HIS PRO TYR TRP SER THR GLY ALA MET CYS PHE LEU VAL ILE
●有機概念図I/O値順(特性基 R)
ASN SER ASP GLN GLU THR ARG HIS GLY LYS TYR TRP CYS MET PRO PHE ALA VAL LEU ILE
●等電点順
ASP GLU CYS ASN PHE GLN TYR SER MET TRP VAL GLY LEU ALA ILE THR PRO HIS LYS ARG
●ヒトの必須・非必須区別
ILE LEU VAL PHE MET TRP LYS HIS THR ARG ALA PRO CYS TYR GLY GLU GLN ASP SER ASN
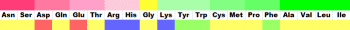
アミノ酸および特性基の親水性・疎水性 | Log Pをポケットに!
初期表示はイチゴの香り物質 (R)-(+)-フラネオール((R)-(+)-furaneol);上掲書p.245
[Claudin - Wikipedia,Enterotoxin - Wikipedia]
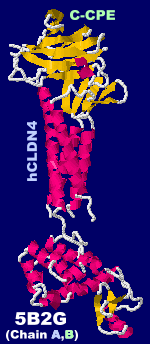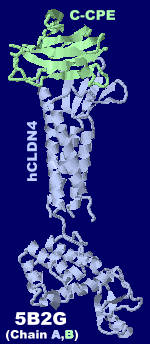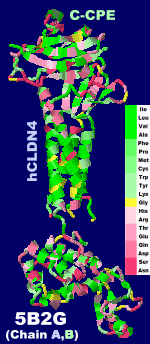
クローディン4とエンテロトキシンC-CPEの複合体5b2gのChain A・B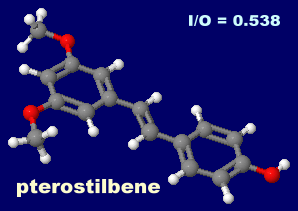
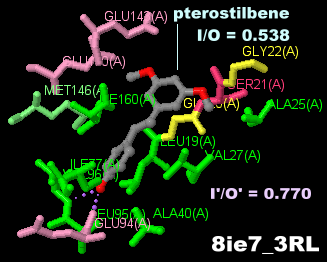
左から プテロスチルベン(pterostilbene),同分子結合データ例の細胞死関連プロテインキナーゼ1(DAPK1)8ie7とPDBsumデータ
[Three-finger toxin - Wikipedia,Cobratoxin - Wikipedia]
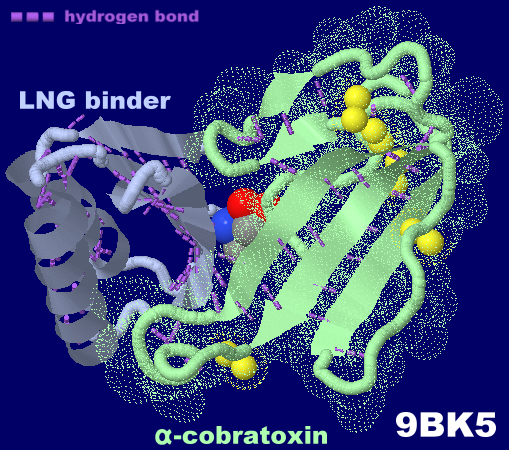
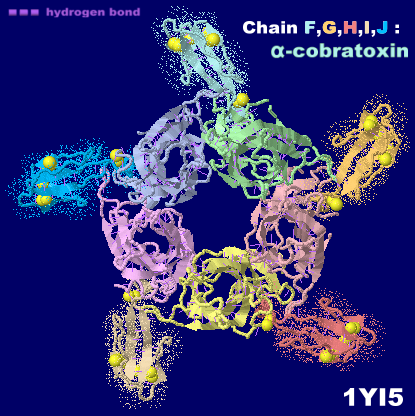
[左]LNGタンパク質とコブラ毒素α-コブラトキシンの複合体9bk5
[右]α-コブラトキシンが結合したニコチン性アセチルコリン受容体1yi5
※何れも黄色球表示はα-コブラトキシン中のS原子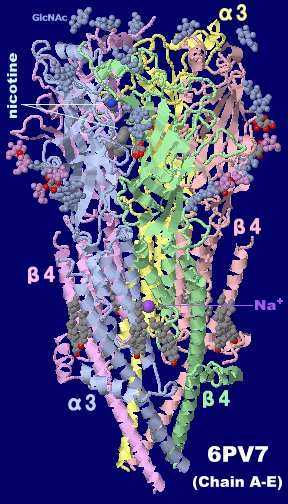
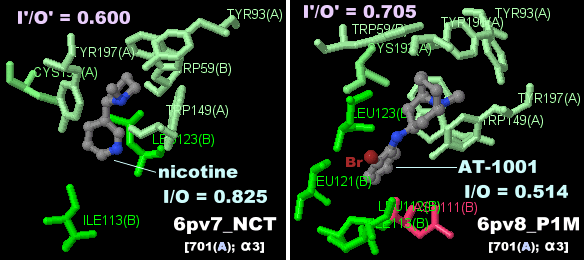
参考図:ニコチンが結合したヒトα3β4ニコチン性アセチルコリン受容体6pv7のChain A-Eと
そのPDBsumデータ(AT-1001結合の6pv8との比較)
→ 別トピック
[U6 spliceosomal RNA - Wikipedia]

U6 snRNAとTUT1の複合体9j8p
I = 230, O = 180, I/O = 1.278
[Gabapentin - Wikipedia]
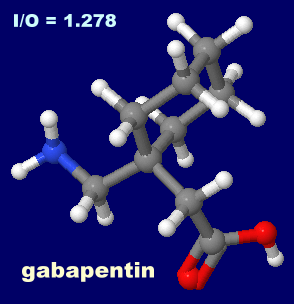
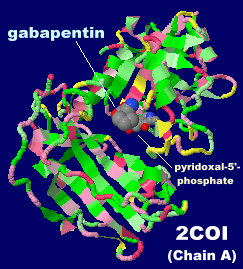
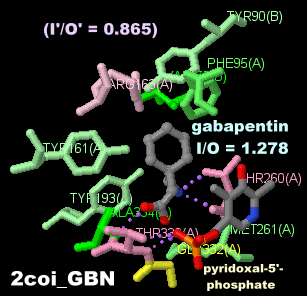
[左]ガバペンチン(gabapentin)
[中・右]同分子が結合した分岐鎖アミノ酸アミノトランスフェラーゼ1(BCAT1)2coiのChain AとPDBsumデータ
[Riboswitch - Wikipedia]
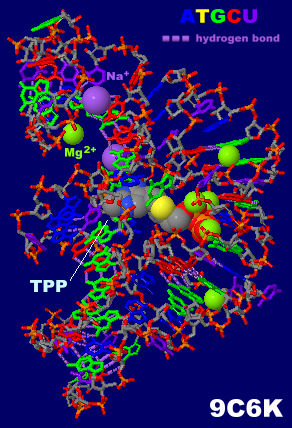
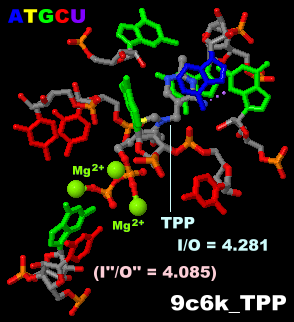
TPPリボスイッチアプタマードメイン(RNA)9c6kとPDBsumデータ
I = 575, O = 445, I/O = 1.292
[Suzetrigine - Wikipedia]
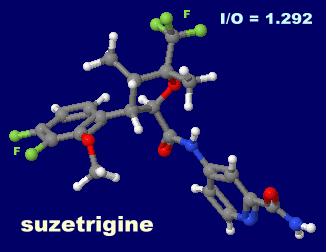
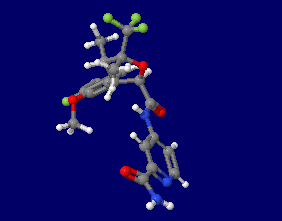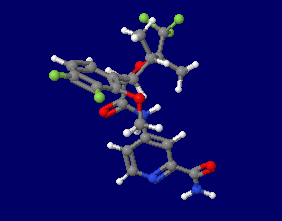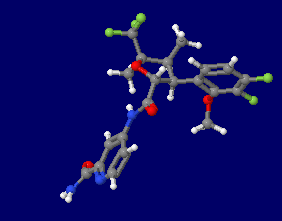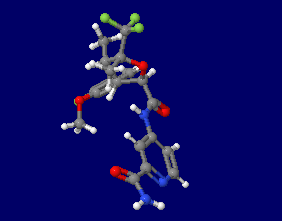
スゼトリギン(suzetrigine)
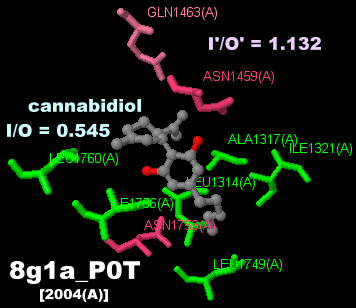
参考図(ナトリウムチャネル構造例):カンナビジオール(cannabidiol,CBD)結合電位依存性ナトリウムチャネル
Nav1.7 8g1aのChain AとPDBsumデータ
→ 危険ドラッグ
講談社作成動画

地学関連書籍を並べて:左隣は「藍を継ぐ海」,右隣は「みんなの高校地学」
(IUPAC名:4-ヒドロキシ-2,5-ジメチルl-3-フラノン,4-hydroxy-2,5-dimethyl-3-furanone〈HDMF〉)
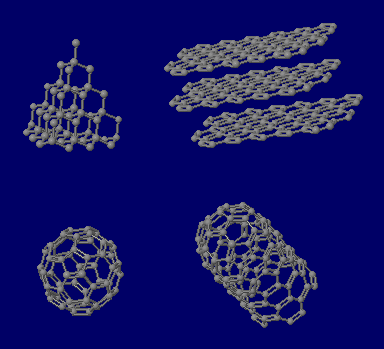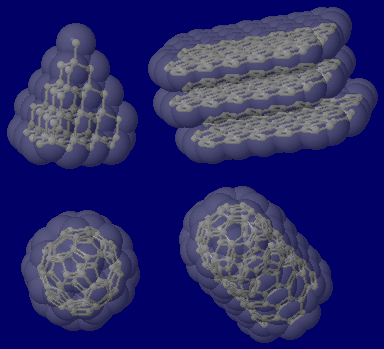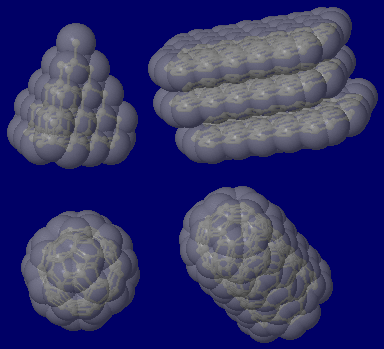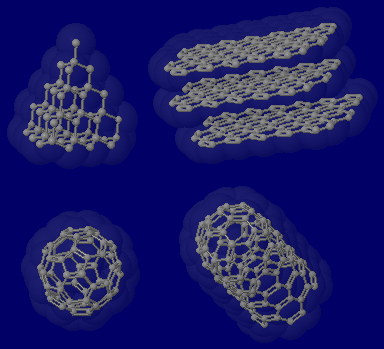
炭素(C)の同素体
ダイヤモンド,黒鉛(グラファイト),フラーレン(C60),カーボンナノチューブ(アームチェア型の例)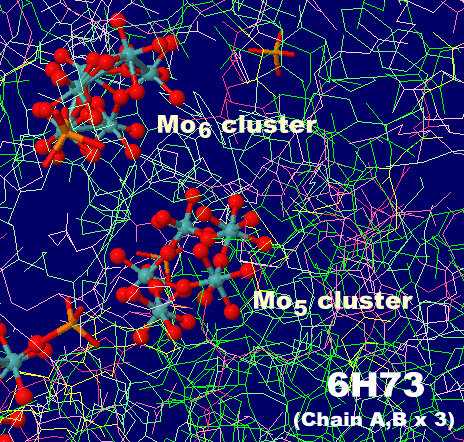
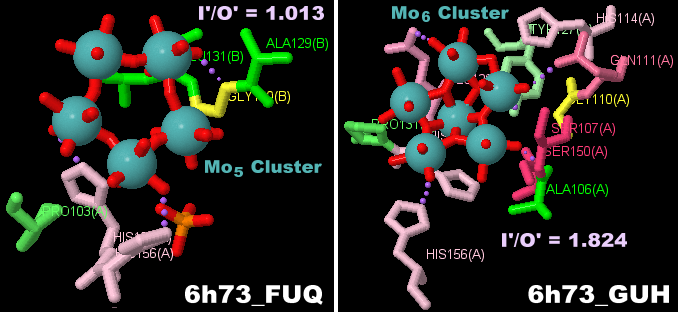
モリブデン貯蔵タンパク質6h73 6量体(Chain A・B×3)のMo5クラスターとMo6クラスター部分の拡大およびそれぞれのPDBsumデータ
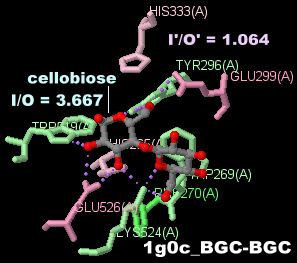
;セロビオースが結合したアルカリセルラーゼ1g0cとPDBsumデータ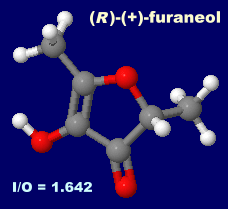
(R)-(+)-フラネオール((R)-(+)-furaneol) → 別トピック
(IUPAC名:4-ヒドロキシ-2,5-ジメチルl-3-フラノン,4-hydroxy-2,5-dimethyl-3-furanone〈HDMF〉)
※フラネオール参考記事例:Periodic Graphics: The Chemistry of Strawberries(Chemical & Engineering News, 2015/06/15)
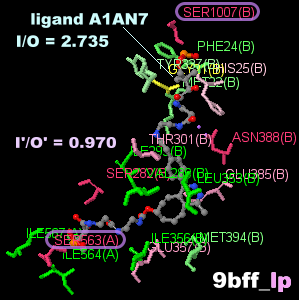
非リボソームペプチド合成酵素9bffとPDBsumデータ
I = 230, O = 380, I/O = 0.605
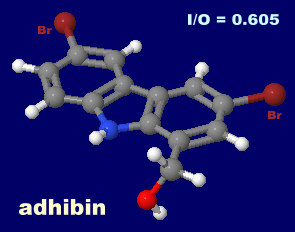
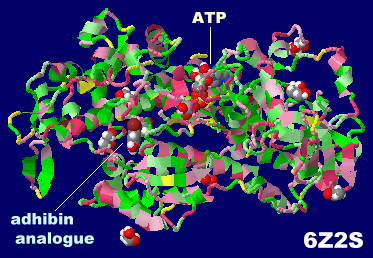

左から アドヒビン(adhibin),アドヒビン類縁体結合のRhoGAPクラスIXミオシン6z2sとPDBsumデータ