�mATP citrate synthase - Wikipedia�n
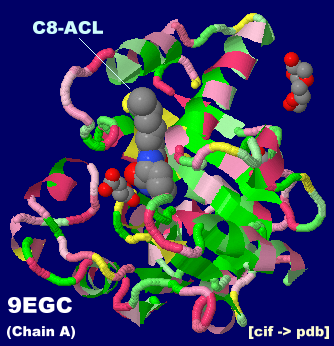
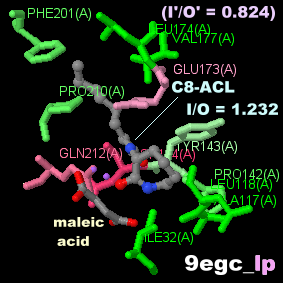
C8-ACL����������AclA�̍\��9egc��Chain A��PDBsum�f�[�^
-
���A�~�m�F�\���̖}��
ASP�@GLU�@CYS�@MET�@LYS�@ARG�@SER�@THR�@PHE�@TYR�@ASN�@GLN�@GLY�@LEU�@VAL�@ILE�@ALA�@TRP�@HIS�@PRO
���_���E�����q�F�����r�E����A�~�m�_��ʕ\���̖}��
ASP�@GLU�@GLY�@ALA�@VAL�@LEU�@ILE�@CYS�@SER�@THR�@ASN�@GLN�@PRO�@MET�@PHE�@TYR�@TRP�@LYS�@ARG�@HIS
���ɐ��q�_���E����r�E��ɐ��i�a�����j�A�~�m�_���
SER�@THR�@TYR�@CYS�@ASN�@GLN�@ASP�@GLU�@LYS�@ARG�@HIS�@GLY�@ALA�@VAL�@LEU�@ILE�@PHE�@PRO�@MET�@TRP
���a�����C���f�b�N�X��
ARG�@LYS�@ASN�@ASP�@GLN�@GLU�@HIS�@PRO�@TYR�@TRP�@SER�@THR�@GLY�@ALA�@MET�@CYS�@PHE�@LEU�@VAL�@ILE
���L�@�T�O�}I/O�l���i������ R�j
ASN�@SER�@ASP�@GLN�@GLU�@THR�@ARG�@HIS�@GLY�@LYS�@TYR�@TRP�@CYS�@MET�@PRO�@PHE�@ALA�@VAL�@LEU�@ILE
�����d�_��
ASP�@GLU�@CYS�@ASN�@PHE�@GLN�@TYR�@SER�@MET�@TRP�@VAL�@GLY�@LEU�@ALA�@ILE�@THR�@PRO�@HIS�@LYS�@ARG
���q�g�̕K�{�E��K�{���
ILE�@LEU�@VAL�@PHE�@MET�@TRP�@LYS�@HIS�@THR�@ARG�@ALA�@PRO�@CYS�@TYR�@GLY�@GLU�@GLN�@ASP�@SER�@ASN
�A�~�m�_����ѓ�����̐e�����E�a���� �b Log P���|�P�b�g�ɁI

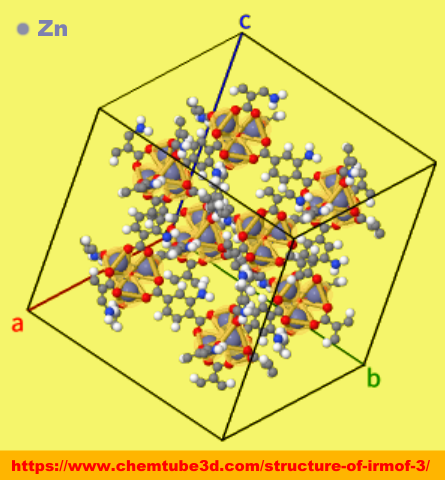 �@���T�C�g�f�[�^�� MOF-5
�@���T�C�g�f�[�^�� MOF-5
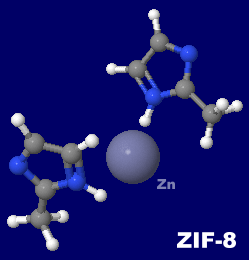
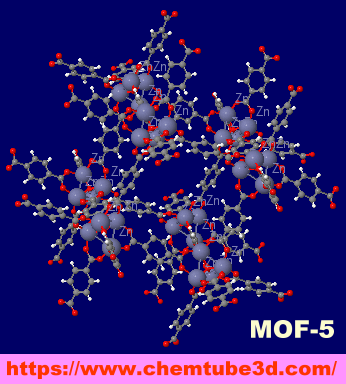
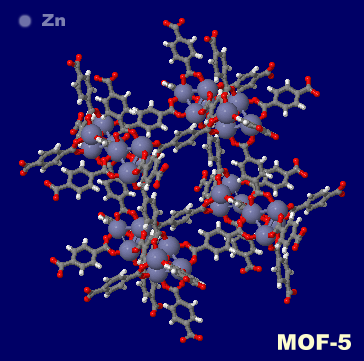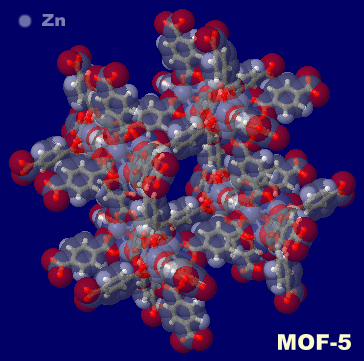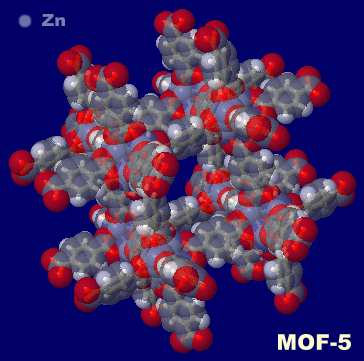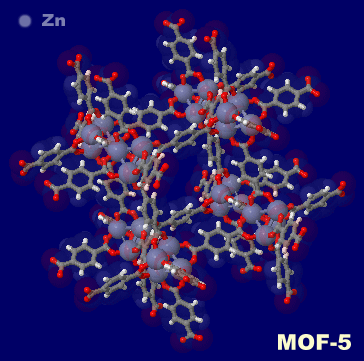
�m���nMOF�\����iZIF-8�C�����\���j
�m���E�E�nMOF�\����iMOF-5�GInteractive 3D Chemistry Animations — ChemTube3D�f�ڃf�[�^��j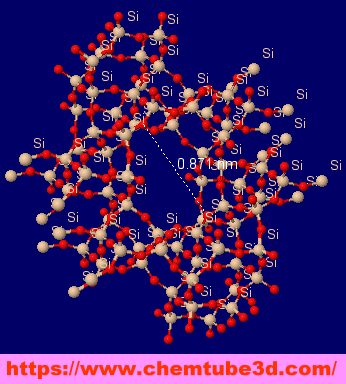
���Q�l�\���i�\�������[�I���C�g�qzeolite�r�����\����FInteractive 3D Chemistry Animations — ChemTube3D�f�ڃf�[�^����j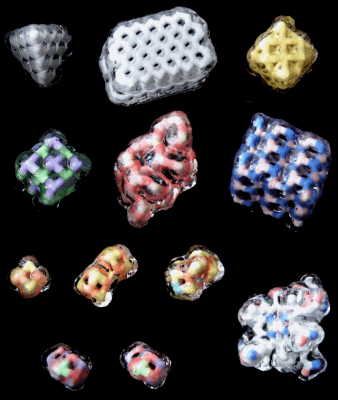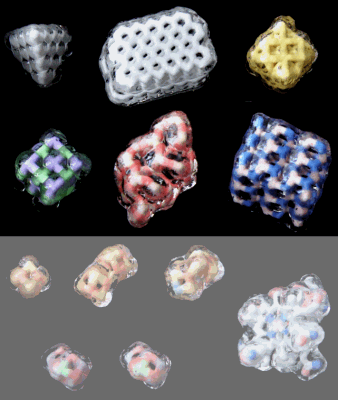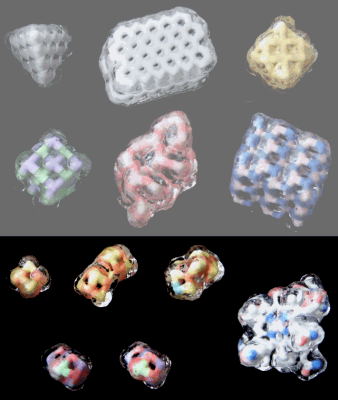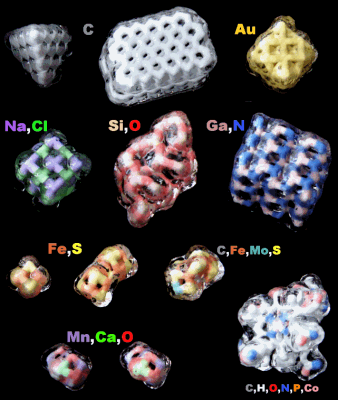
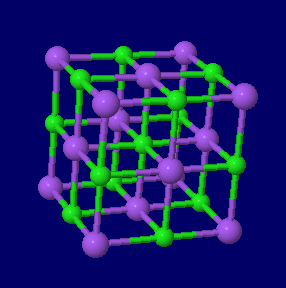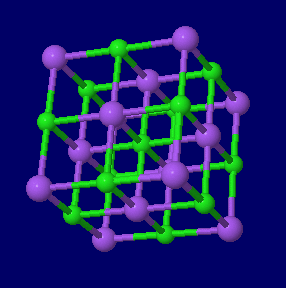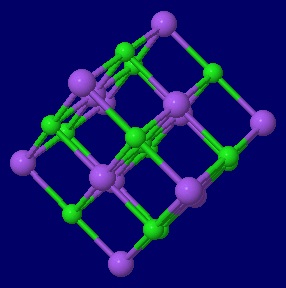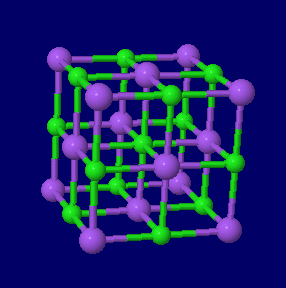

���Q�l�i�ʏ�̌����\����j
������ �e�팋���\��������ヂ�f���C�����i�g���E���isodium chloride�CNaCl�j�C���iAu�j�E��iAg�j�E���iCu�j�̌����\�������\��
��2�҂ɂ��Ắ@�� �ʃg�s�b�N
I = 690, O = 560, I/O = 1.232
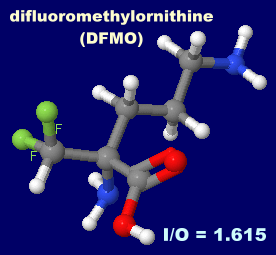
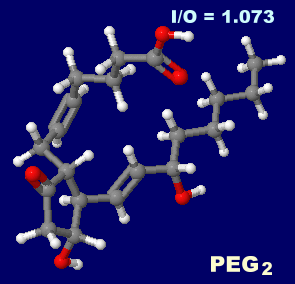
�m�E�nDFMO�@�m���n�v���X�^�O�����W��E2�iPEG2�j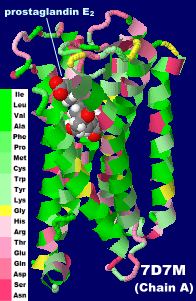
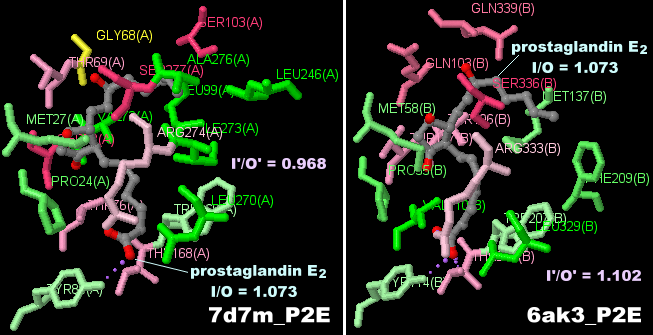
���Q�l�F�m���E���nPEG2����EP4��e�̗�7d7m��PDBsum�f�[�^�i�E���͕ʌ����G���ƌ����͌^�Q�Ɓj
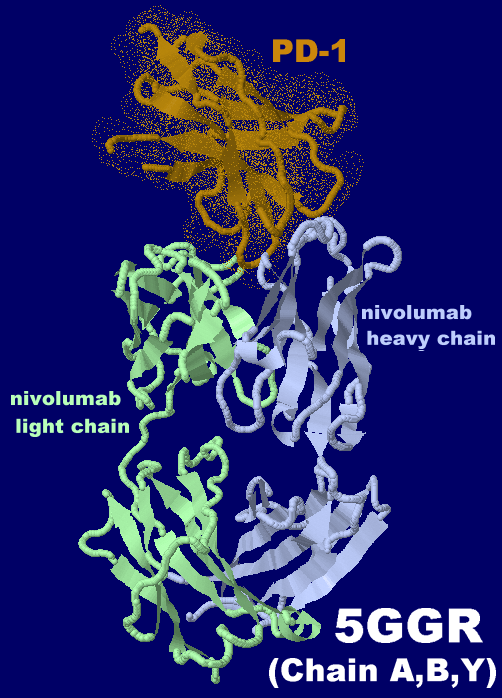
�m�E�n�j�{���}�u�i���i�� �I�v�W�[�{�j��Fab����������PD-1 5ggr�@�� �ʃg�s�b�N
I = 1280, O = 640, I/O = 2.000
�mElamipretide - Wikipedia�n
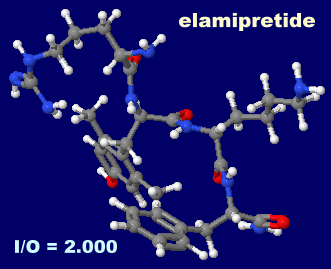
�G���~�v���`�h�ielamipretide�j
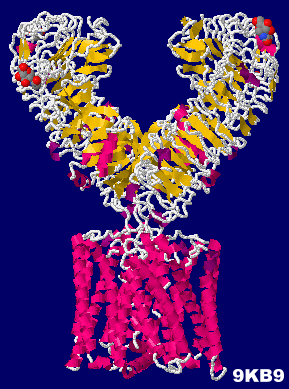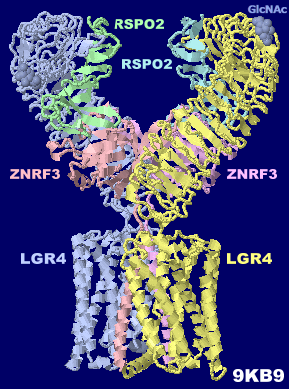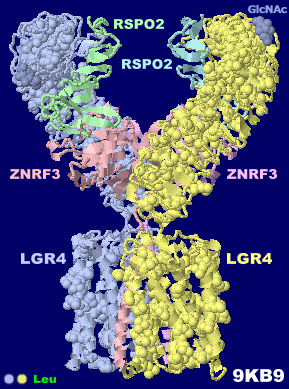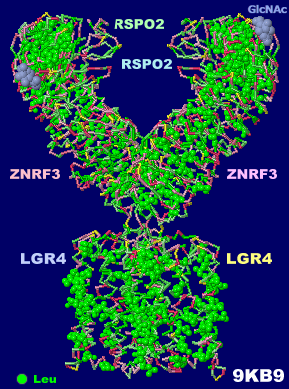
LGR4/RSPO2/ZNRF3������9kb9
I = 210, O = 130, I/O = 1.615

�W�t���I�����`���I���j�`���idifluoromethylornithine�GDFMO�j
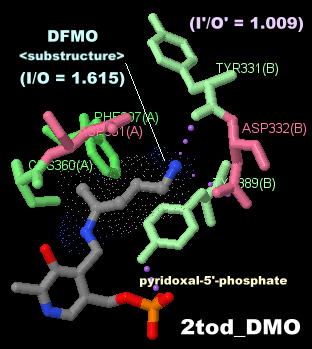

������ DFMO�i�����\���j�����������I���j�`���f�J���{�L�V���[�[2tod��Chain A�EB��PDBsum�f�[�^�C
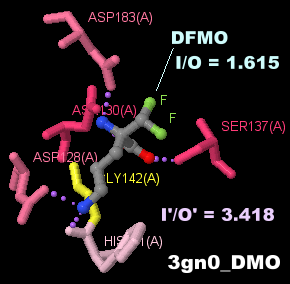
DFMO�����������A���M�i�[�[3gn0��Chain A��PDBsum�f�[�^
I = 227, O = 460, I/O = 0.493
�mRotenone - Wikipedia�n
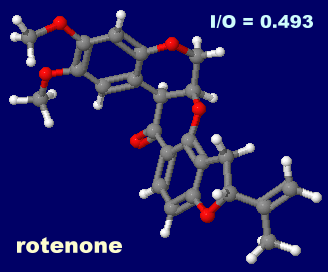
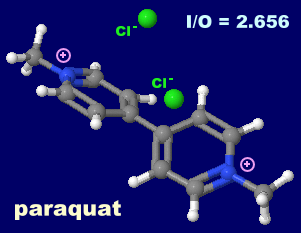
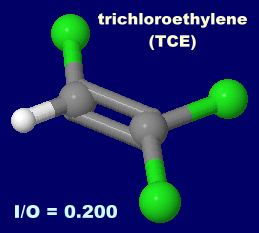
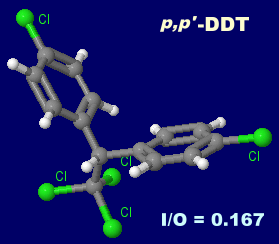
������ ���e�m���irotenone�j�C�p���R�[�g�i�p���N�A�b�g�Cparaquat�j�C
�g���N�����G�`�����itrichloroethylene�CTCE�j�Cp,p'-DDT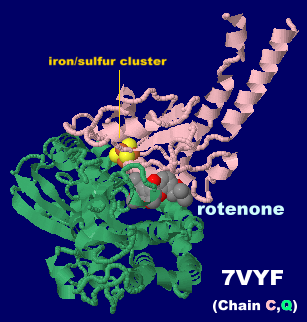
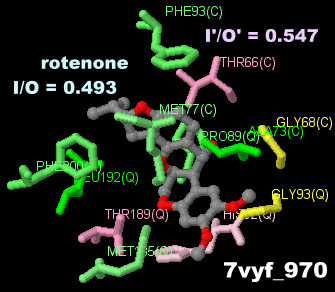
���e�m���irotenone�j�����������~�g�R���h���A������I 7vyf��Chain C�EQ��PDBsum�f�[�^
I = 880, O = 220, I/O = 4.000
�mN-Acetylneuraminic acid - Wikipedia�n
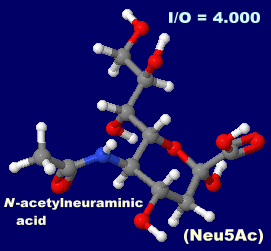
������ N-�A�Z�`���m�C���~���_�iN-acetylneuraminic acid�GNeu5Ac�j�C
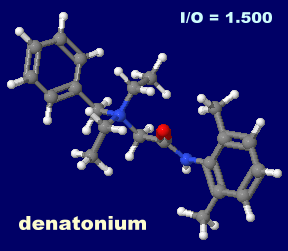
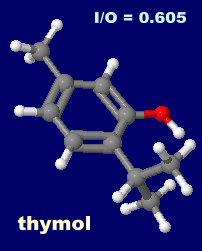
�f�i�g�j�E���idenatonium�j�C�`���[���ithymol�j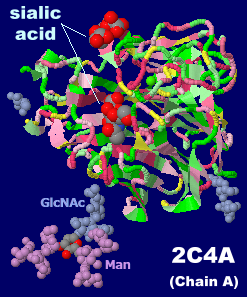
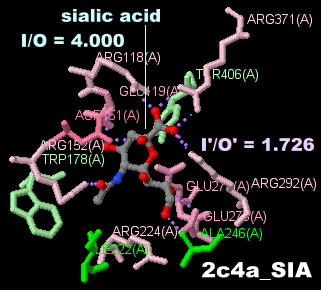
�V�A���_����������A�^�C���t���G���U�E�C���X�iN9�j�̃m�C���~�j�_�[�[2c4a��Chain A��PDBsum�f�[�^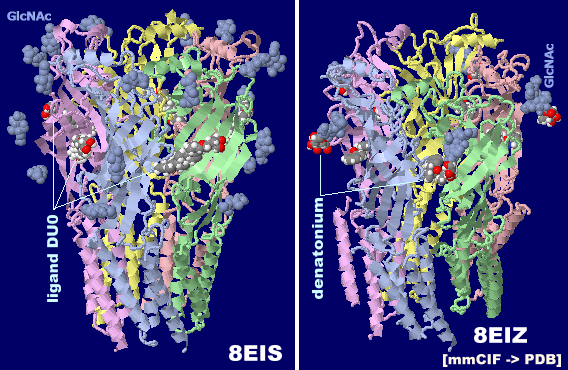
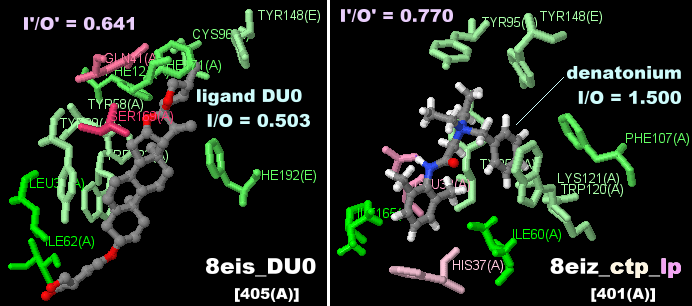
�^�R�̑�������e��CRT1 8eis�ƃC�J�̉��w�G�o��e��CRT1 8eiz�i�f�i�g�j�E���qdenatonium�r�����j�C����т��ꂼ���PDBsum�f�[�^
I = 165, O = 180, I/O = 0.917
�mPhenylpropanoic acid - Wikipedia�n
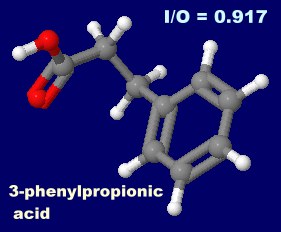
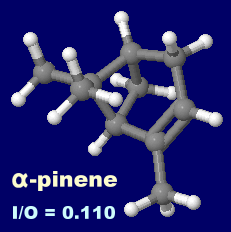
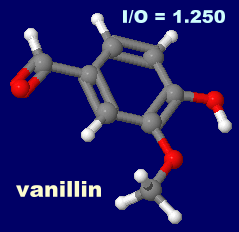
�����҂̍��萬����i������j 3-�t�F�j���v���s�I���_�i3-phenylpropionic acid�j�C
��-�s�l���i��-pinene�G���u�_�i���̐�����j�C
�o�j�����ivanillin�G�o�j���̍��萬����j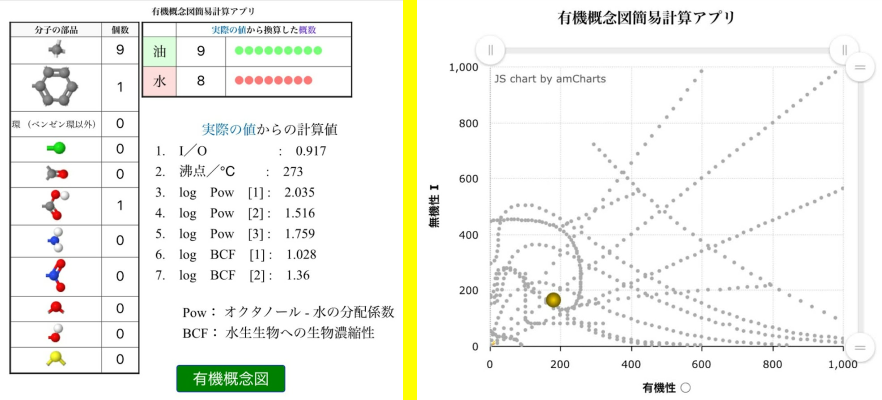
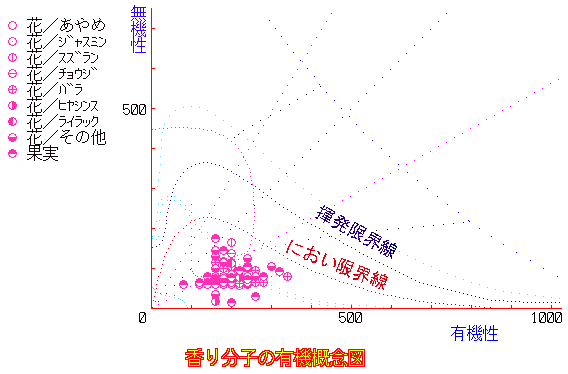
�m�E�n3-�t�F�j���v���s�I���_�̗L�@�T�O�}�i���_��log P�̕����l�͂Ȃ��j
�m���n���蕪�q�̗L�@�T�O�}
I = 280, O = 405, I/O = 0.691
�mMidazolam - Wikipedia�n
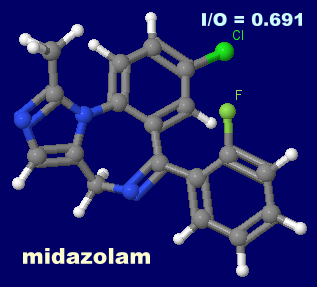
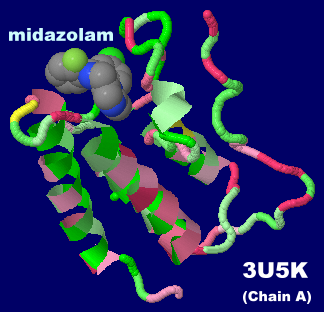
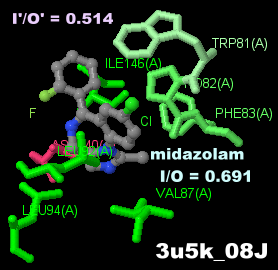
������ �~�_�]�����imidazolam�j�C�����q����BRD4�̍\��3u5k��Chain A��PDBsum�f�[�^